info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
三代测序(PacBio)
PacBio三代测序——同一平台可获得高质量基因组,表观遗传学(甲基化修饰),全长转录组,染色质可及性等多组学的数据,核心是精准长读长测序,即HiFi测序。
产品介绍
PacBio三代测序——同一平台可获得高质量基因组,表观遗传学(甲基化修饰),全长转录组,染色质可及性等多组学的数据,核心是精准长读长测序,即HiFi测序。在此基础上,可进行NGS测序无法涉及的各种变异检测,转录本可变剪切分析,转录本水平的差异表达及功能注释,表观遗传学分析,染色质可及性分析,长距离定相(phase)等多个组学分析以及联合分析。
技术优势
① HiFi测序:高准确性(单分子准确度≥99.9%)、长读长(可达25 kb)、单分 子分辨率、无GC偏好。
② 可均匀覆盖基因组上富含G/C、重复、高同源区域等二代测序无法覆盖的区域, 并获得位于这些区域上被遗漏的变异,可获得更完整的基因组并解释一些位于此区 域上的致病变异导致的疾病病因。
③ 全面覆盖各种变异类型:除了小的单碱基变异及插入缺失,可以覆盖更多的结 构变异,并获得变异定相信息,通过判断这些变异是否位于同一等位基因上还是两 个等位基因上来判断其是否失活或是携带者,提高对疾病病因的诊断。
④ 无需二次建库和重亚硫酸盐处理,获得基因序列的同时,获得定相甲基化修饰 信息信息。
⑤ 可实现单倍型分辨率的基因组组装及泛基因组构建。
⑥ 无需拼接,可通过全长RNA测序实现更全面的转录组分析
⑦ 50000+文献支持。
⑧ 专业的技术支持及生信分析团队:国内率先成立的三代测序平台专业技术支持 团队,秉承十多年的支持经验,为用户提供高品质的实验服务和方案,挖掘更多数 据价值。
样品类型
1新鲜冰冻或FFPE组织 ;2培养细胞; 3血液样本;
标准结果交付
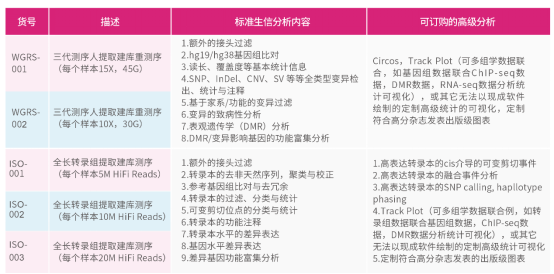
l 人基因组重测序:实验报告,质控报告,原始数据和分析报告。
l 全长转录组测序(ISO-Seq):实验报告,质控报告,原始数据和分析报告
订购信息