info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
细胞样本中病毒滴度检测和抗病毒药物筛选,选用 In-cell western 技术就对了

对于研究病毒的科研人员来说,了解病毒的感染特性是至关重要的。例如引起COVID-19的冠状病毒SARS-CoV-2,标准菌斑分析法(SPA) 是研究病毒感染特性和病毒滴度的传统方法。但该方法存在一定的局限性,操作繁琐,通量低,无法准确定量,并且容易引入认为误差。
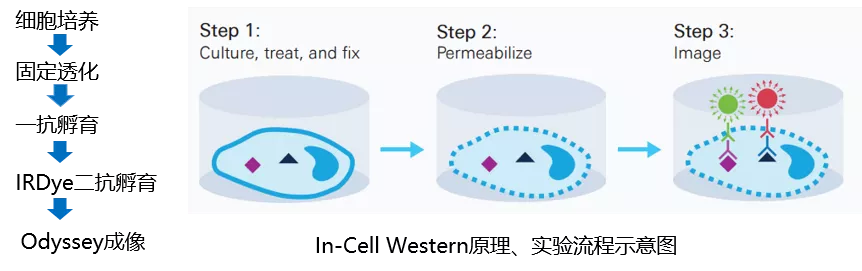
In-Cell Western技术,是一种依据抗原抗体结合原理,利用目标蛋白的特异性抗体及近红外荧光染料标记的二抗,在96孔板或384孔板培养的细胞上原位对目标蛋白进行定量分析的技术。该技术结合了 Western blot 技术的特异性与 ELISA 技术的可复制性和通量,目前已成为研究病毒感染特性的高效、快速、可靠的实验技术。
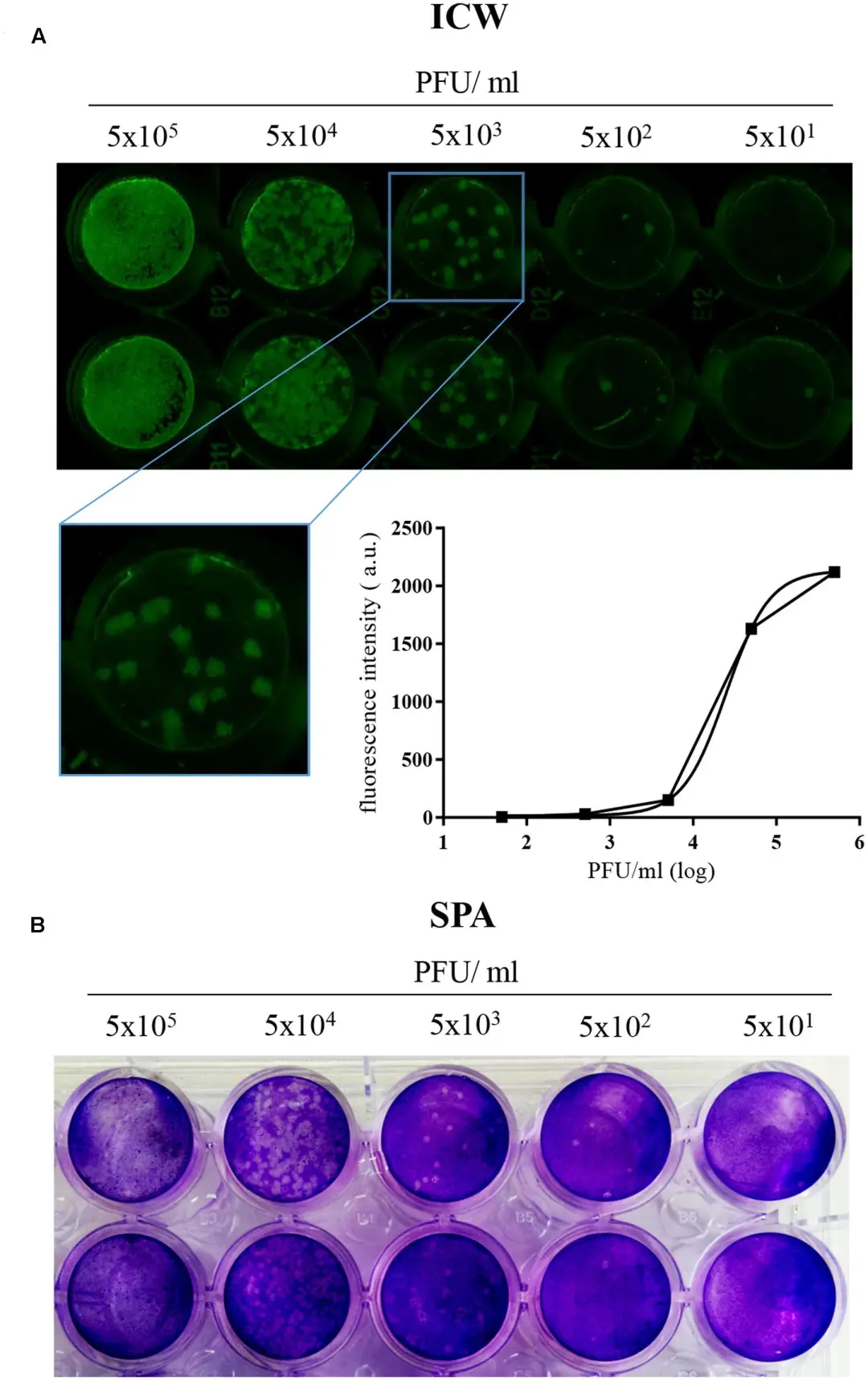
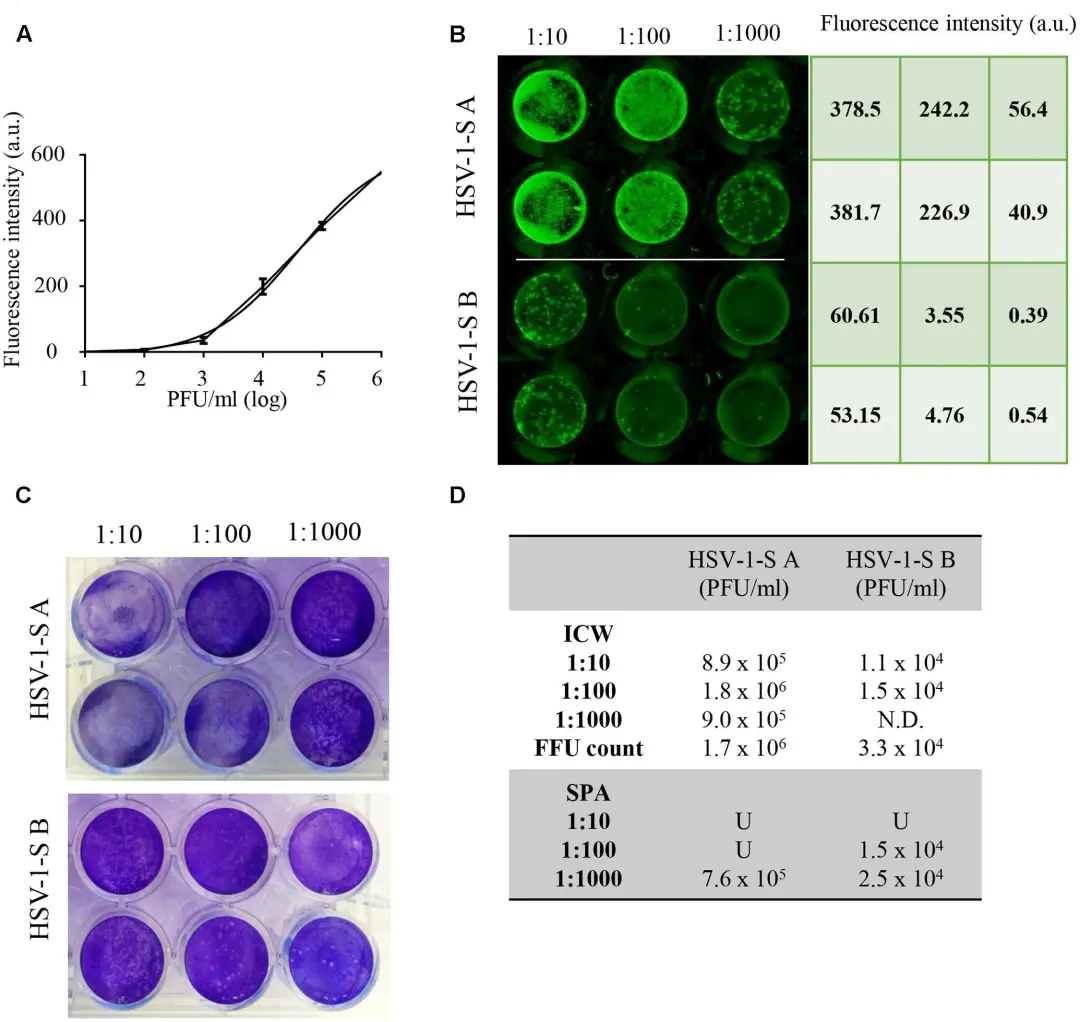
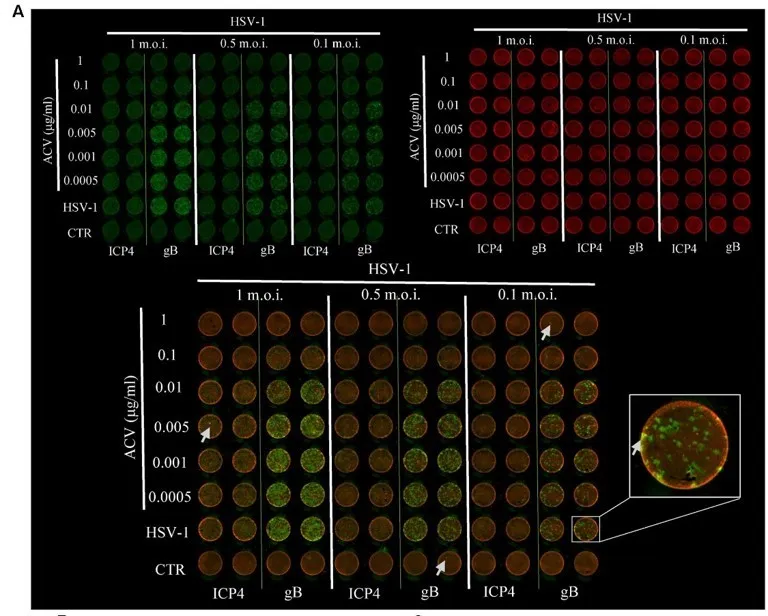
In-cell western 技术原理与实验流程 In-cell western 技术可监测细胞感染的HSV-1病毒 在96孔细胞培养板中用不同滴度的 HSV-1 病毒感染 Vero 细胞,完成细胞固定和一抗孵育后,使用LI-COR 近红外荧光二抗孵育,更后用 Odyssey 近红外成像系统检测荧光信号。不仅可以通过荧光信号强度判断细胞所感染的病毒滴度,而且可以根据荧光信号的位置锁定病毒感染部位。标准菌斑分析法(SPA)佐证了 In-cell western 实验结果的准确性 。 In-cell western 技术可对细胞感染的 HSV-1 病毒进行准确定量 SPA 法仅可对较低滴度的 HSV-1 感染的细胞中的病毒进行粗略定量,且需要稀释病毒,误差较大,过程繁琐,通量低,对于较高滴度的病毒无法计数。In-cell western 技术则可直接对感染高滴度病毒的细胞中的HSV-1 进行检测,无需稀释,通量高,速度快,定量准确。 In-cell western 技术适用于抗 HSV-1 病毒药物的筛选 预先使用不同浓度梯度的抑制HSV-1病毒复制的ACV药物处理Vero细胞,然后用 0.1, 0.5 和 1m.o.i. HSV-1 感染细胞,通过对病毒感染早期的ICP4 蛋白和感染晚期的gB蛋白的定量来判断ACV药物的效果,Cell-Tag 700 荧光染料对细胞进行染色起到均一化细胞数量的作用。 In-cell western 技术优势 省时,直接在微孔板检测,无需细胞裂解、跑胶和转膜等过程 高通量,一次可实现96/384个样品检测 数据变异小,直接对细胞内的蛋白进行检测,特异性好,灵敏度高 双通道同时检测,不同荧光标记二抗同时检测两个蛋白 In-cell western 应用广泛 高通量快速的In-Cell Western已广泛应用到细胞自噬、凋亡、癌症、药物研究、信号转导、离子通道、基因表达等多个方面。更多应用文献请登录 LI-COR 官网查询:https://www.licor.com/bio/publications/ 参考文献: 基因有限公司作为LI-COR近红外成像解决方案的合作伙伴与中国区独家代理商,与LI-COR一起邀请您一同关注近红外荧光成像技术在Western Blot准确定量、磷酸化等蛋白修饰化研究、In-Cell Western、双色EMSA、蛋白芯片及光免疫疗法等方面的应用进展。欲进一步了解详细内容,请关注我们的官方微信“基因快讯”或联系您身边的基因有限公司工作人员。
Weldon, S.K., et. al. (2010). Journal of Virological Methods, 168(1–2), 57–62. DOI: 10.1016/j.jviromet.2010.04.016
Fabiani, M., et. al. (2017). Frontiers in Microbiology, 8(1085). DOI: 10.3389/fmicb.2017.01085
DuShane, J.K., et. al. (2019). Frontiers in Microbiology, 10(783). DOI: 10.3389/fmicb.2019.00783
Ma, H. W., et. al. (2017). Frontiers in Cellular and Infection Microbiology, 7(269). DOI: 10.3389/fcimb.2017.00269
McCormick, D. et. al. (2018). mBio, 9(3), e00716-18. DOI: 10.1128/mBio.00716-18
Sklan, E. H. et. al. (2007). Journal of Virology, 81(20), 11096–11105. DOI: 10.1128/JVI.01249-07