info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
在SMRT测序中应该怎样提升HiFi reads组装的连续性?

近日,中国科学家在冷泉港的文章预印网站bioRxiv上发表了有关PacBio HiFi reads与Nanopore ultralong reads在水稻基因组组装效果比较的文章,“Comparison of the two up-to-date sequencing technologies for genome assembly: HiFi reads of Pacbio Sequel II system and ultralong reads of Oxford Nanopore.”。发现来自Nanopore的数据由于错误率高,使得近1500个基因的注释错误;相对应的,无论是在SNP还是小的indel的读取上,PacBio HiFi reads则具有更高的准确度。
不过,或许您对于这篇基因组组装比较的文章,还有更多有关PacBio HiFi reads的疑问,相信在了解以下内容后,您会对HiFi reads在基因组组装应用方面有更深入的了解。
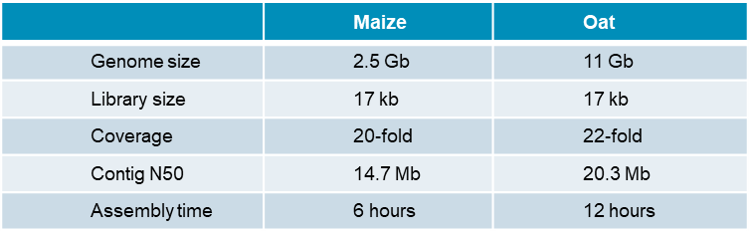
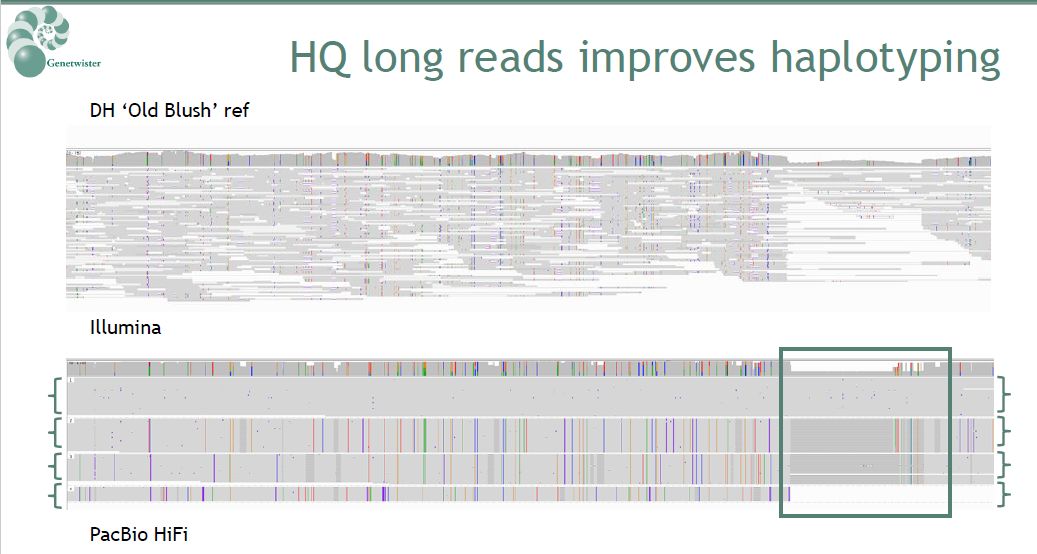
首先 01 什么是PacBio HiFi reads? PacBio的单分子实时(SMRT)测序技术经过多年的发展,已经使酶读长度增加到平均100 kb。通过优化和平衡酶读长与插入片段的长度,开发了一种基于CCS(Circular Consensus Sequencing)的高准确率长读长测序模式——HiFi Reads。能够在提供长读长reads(10-20kb)的同时兼顾准确性(Q30,99.9%准确率)。HiFi Reads的生成原理请参考下面动图。 图1:PacBio HiFi Reads的生成原理 点击链接,了解有关PacBio HiFi更详细的内容:Nature Biotech文章验证了PacBio HiFi Reads进行基因组组装及变异检测的出色性能 读长 02 文章中为何HiFi reads没有带来更大Contig N50 由于此次PacBio HiFi reads,来自于作者采用过短的11 kb插入片段来构建的文库,在组装的结果中,相较而言Nanopore ultralong reads在组装具有更强的连续性。 在2019年Sequel II系统正式面世后,基于Sequel II平台的相关试剂、软件都有了更进一步的提升。目前在Sequel II上,能够支持的HiFi文库构建已经扩大至20 kb。更大的插入片段构建的文库,更大程度的兼顾了PacBio超长读长的发挥,以及高度准确性。无论是跨越重复序列,还是序列中微小的差异,甚至是那些位于重复序列中的微小差异都能够很好的识别。事实上,在比水稻基因组更大的物种,例如玉米(2.5 Gb),燕麦(11 Gb)中,这一优势已经已经得到了证实。甚至,四倍体玫瑰(600 Mb x 4)的基因组组装也可以通过HiFi reads得到基因组中等位基因准确的单倍型结果。 表1:HiFi reads在大型基因组中的应用表现 图2:HiFi reads组装四倍体玫瑰基因组 您可以点击文末的“阅读原文”,了解2020年PAG大会上四倍体玫瑰基因组的报告分享 在bioRxiv预印网站该文章的下方,社交平台对文章的讨论中,来自PacBio的专家Elizabeth Tseng也提出,以文章中采用的11 kb来进行HiFi reads的组装分析,往往是不够的。通常需要15 kb以上的文库,才能更好的解决重复序列的问题。这一观点也得到了不少学者的认可。 分析 03 PacBio HiFi reads在分析方面有什么优势? 另一方面,由于HiFi reads已经完成了单分子的纠错,因此在组装过程中可以大大简化流程,与CLR(Continuous Long Read)模式相比,可大大节约组装所消耗的时间。并且,还可以支持等位基因单体型的组装。上述提到的四倍体玫瑰项目,就获得了不同单倍型的准确结果。另外,目前,也已经有不少针对HiFi reads开发的基因组组装软件,包括HiCanu,Hifiasm,补充了HiFi流程的Falcon等等,都可以作为基因组组装分析的优选工具。 在bioRxiv预印文章的讨论区,PacBio资深专家Armin Töpfer 博士向作者推荐了更新版本的Canu。有关如何使用HiCanu,也可以参考下方链接中的内容。 https://github.com/marbl/canu/issues/1601 成本 04 PacBio HiFi reads数据产出及芯片成本? PacBio HiFi reads在数据量的产出方面,经过2019年2.0版本试剂的升级,一张SMRT Cell 8M芯片更高可产出500 Gb的数据,经CCS分析后,可获得超过25 Gb准确率在Q20以上的数据,用于更准确的组装分析。相对于原来的CLR方式,HiFi reads的覆盖深度也有可降低,在15-20X左右即可获得高质量的基因组组装结果。因此,即使对于本文所采取的水稻作为研究对象,一张SMRT Cell 8M芯片即可完成高质量的基因组组装,并能够对基因组中包括SNP及结构变异在内的所有变异进行检测。 综合以上内容,不难看出。目前已经可以采用更优化的手段运用HiFi reads来进行基因组的组装与分析。无论是在分析软件的选择,还是数据产出与成本控制方面,已经有了大幅的改善。目前20 kb的文库插入片段,已经可以更加完美的兼顾读长与准确度。相信对于绝大多数研究领域而言,PacBio HiFi reads带来的更为准确度的基因组组装结果,将会是进行更深入研究的良好基础。 参考原文: Lang, Dandan, et al. "Comparison of the two up-to-date sequencing technologies for genome assembly: HiFi reads of Pacbio Sequel II system and ultralong reads of Oxford Nanopore." bioRxiv (2020). 基因有限公司作为PacBio公司在中国区的独家代理商,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。