info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
使用DS3000 Compact CE Sequencer提高短DNA读取精度

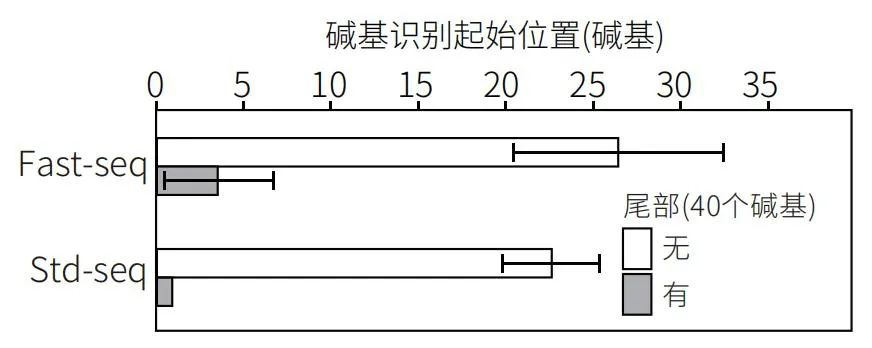
“概要” 在Sanger测序中,时有在开始读取的区域中无法获得峰波形,或者出现读取错误的情况。这是由于填充在毛细管中的电泳介质 (聚合物) 有无法正确检测,分离短DNA的情况。 此实验是使用DS3000 Compact CE Sequencer,提高了开始读取区域的数据质量[1] 的示例。与以往不同之处在于使用了加尾引物,测序引物5’末端加入任意碱基序列延长反应产物。用处在于当添加40个碱基至尾部时,可从测序引物的3’末端下游第1个碱基的DNA开始无错误地读取。 另一方面,由于尾部碱基的添加,碱基有效读取的长度也相应缩短。因此在添加尾部碱基时,请参考本报告,事先确认分析区间。 “结果” 对质粒DNA进行测序 使用T3引物对质粒DNA(pTS1) 进行了测序。测序引物尾部添加了40个碱基。 首先,通过峰波形确认了加尾引物的效果(图1,请注意图中的碱基长度是以引物的3’末端为0计数的)。在没有尾部引物的测序结果中,无法确认从引物的3’末端到第17个碱基的峰波形。第20个碱基以后可以确认明确的峰波形,但仍发生了读取错误(图1A红框部分)。 相反,在加尾引物的测序结果中,可以确认到从引物的3’末端开始的第1个碱基的峰波形。并且,在图中的范围内未发生读取错误。 图1. 峰波形比较 显示了使用无尾引物(A)与加尾引物(B)得到的峰波形。碱基长度是将引物的3’末端为0来计数。请注意与普通的测序查看器不同。 红色阴影处表示获得与正确序列不同结果的部分。使用Std-seq实施测序方式。 碱基识别起始位置的比较 其次,比较了碱基识别的起始位置(图2)。 DS3000有2种测序模式。通常,Fast sequencing (Fast-seq) 适用于在短时间内得到尽可能长的读取长度,而Standard sequencing(Std-seq)适用于需得到比Fast-seq更长的读取长度的情况。 在本实验中,分别使用两种测定模式进行了验证。结果显示无论使用哪种测定模式,均可通过添加尾部,获得准确的碱基识别的峰波图。特别是在使用Std-seq的情况下,16次实验均从第1个碱基开始准确地进行碱基识别。 图2. 碱基识别起始位置的比较 显示了以测序引物的3’末端为起始,计数从第几个碱基开始DS3000开始了准确的碱基识别。在每个条件下进行了16次实验。 柱状图表示平均值,bar表示标准偏差。在Std-seq中,因为有加尾引物的存在,所有实验均从样本第1个碱基开始碱基识别,所以没有bar。 序列读取精度的比较 最后,比较了准确读取结束的位置。图3显示了识别出碱基的准确性保持在99%以上的最后一个碱基的位置。在两种测定模式中,加尾均缩短了读取结束位置(图3)。 作为电泳的特性,DNA越长,分离性能越低,更难正确识别碱基。加尾引物添加得到碱基使得样本碱基起始得以准确识别,同时占用了一定的读取长度,因此长碱基的分离性能将降低,准确读取结束位置相比无加尾引物会提前。 图3. 读取碱基结束位置的比较 显示了在各条件下获得的读取结束位置。读取结束位置显示了识别的准确性维持在≥99%的最后的碱基的位置。 以测序引物的3’末端为0来计数。柱状图为16次实验获得的平均值,bar表示标准偏差。使用威尔科克森检验计算p值。 “结论” 将40个碱基添加到测序引物尾部,可改善开始读取区域的读取错误与碱基识别的起始位置。特别是在使用Std-seq的验证时,在所有实验中,从引物的3’末端下游的第1个碱基开始获得清晰的波形数据与准确的碱基识别。 另一方面,当添加尾部时,读取的碱基长度将相应变小。在添加尾部时,请参考本报告,事先确认分析区间。 注意事项 当测序引物的尾部与模板DNA退火时,测序数据中会出现噪音并降低准确性。使用软件设计尾部的碱基序列时,应尽量避免其与模板DNA发生配对,要特别注意尾部与模板DNA退火区域的Tm值,应不超过引物的Tm值。避免引物二聚体或发夹结构[2],且尾部不宜太长,建议一般≤40个碱基。 “实验方法” 将pTS1作为模板进行循环测序(表1~3)。反应产物通过乙醇沉淀纯化,并溶解在10 μL HiDi formamide (Thermo Fisher Scientific)中。使用DS3000进行测序分析(表4)。峰波形是基于sangerseqR3绘制的。





参考文献 1.Binladen, J., et al. 5’-Tailed sequencing primers improve sequencing quality of PCR products. BioTechniques. 42 (2), 174-176 (2007) 2.Untergasser, A., et al. Primer3̶new capabilities and interfaces. Nucleic Acis Research. 40 (15), e115 (2012) https://www.primer3plus.com/ 3.Hill, J., T., et al. Poly peak parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243, 1632-1636 (2014) · 本资料中刊登的数据仅为测试示例,不确保数据的准确性。 · 本资料中的图片为嵌入合成图。 · 该系统仅供科研使用,不用于任何动物或人类的治疗或诊断用途。 · 本资料中的公司名称、商品名称为各公司的商标及注册商标。

关于基因 基因有限公司成立于1992年,是一家提供生命科学科研仪器、试剂耗材和技术服务的综合服务商。基于“Gene Brightens Every Life • BioTech Connects the World”——“基因燃亮生命 • 生物技术连接世界”的愿景,专注于生命科学领域前沿技术的引进和推广,致力于推动该领域国内科研机构硬件水平及实验方案的革新与升级。同时,公司也一直致力于自主品牌的科研设备的研发与生产,拥有一系列通用性强、互补性高的自主品牌产品。 基因的服务网络遍及全国各地十多个大中城市,拥有包括仪器销售,试剂销售,市场与技术支持,维修,客服,物流等多个部门组成的完整服务体系。 我们希望通过不懈努力,为您的成功铺路搭桥,也为中国的生命科学事业赶超世界先进水平尽一己之力。欲了解更多信息,请访问www.genecompany.cn。