info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
长读长测序揭示癌症基因组中端粒相关的复杂变异

12 端粒是基因组中由短的,多重复的非转录序列(TTAGGG)及一些结合蛋白组成特殊结构,其结构和位置的改变是癌症基因组进化的关键事件。然而,过去很长一段时间,基于短读长的NGS技术无法跨越基因组中长端粒的重复阵列,继而也无法表征这些关键的改变。在长读长测序技术,特别是高精准的PacBio HiFi测序问世后,癌症和癌细胞系中含有端粒重复的结构将重新得以准确评估。
在过去十五年,高通量的NGS发展极大地加快了我们对癌症基因组的表征,如今我们能够对数万个样本中的体细胞变异和遗传性变异进行详细而快速的表征,这也使得我们直接发现了许多癌症驱动基因的改变,并使之成为新兴疗法的潜在目标。 尽管基因组测序技术取得了重大的进步,但约占人类基因组的一半的重复元件,在体细胞中改变的识别和表征仍然是重大挑战。端粒是高度重复结构的一个突出例子,在癌症中尤为重要,但目前的测序方法尚无法轻易解决这一问题。人类端粒作为染色体末端的保护帽,由 2-10 kb(TTAGGG)的串联重复序列组成。肿瘤样本中还观察到端粒序列整合至非端粒 DNA 的体细胞变异,但这些序列的起源和结构仍不清楚。由于通常采用的 NGS 短读长测序技术(如 2 x 150 bp )无法完全跨越 2 kb – 10 kb 长的高度重复端粒,因此癌症中的端粒结构仍有很多未知之处。
近日,美国波士顿 Dana-Farber 癌症研究所以及哈佛医学院等单位共在bioRxiv上共同发表了一篇文章,基于包括PacBio HiFi 以及Nanopore的长读长和NGS短读长测序技术,来评估癌症和癌细胞系中含有端粒重复的结构。
01 长读基因组测序揭示癌症中的新端粒
长读长测序分析表明,标准方向的异位端粒重复序列很长且无界,因此与新端粒添加一致。例如,在短读长测序数据中,观察到与 U2-OS 骨肉瘤细胞系中的 chrX:103,320,553 序列相邻的候选异位端粒重复序列位点包含至少 7 个串联(TTAGGG)重复序列(Fig 1A),以及该染色体位置处对应于端粒重复位置的测序覆盖度的减少(Fig 1B)。而在PacBio HiFi 和 Oxford Nanopore的长读长测序数据集中,通过分析该区域,可以很容易地观察到标准方向上约 3-10kb 的长端粒重复(Fig 1C),其中读长之间端粒长度的变化可能与可以用 DNA 复制后活跃的端粒序列丢失或端粒维持来解释。

Fig.1 通过长读基因组测序揭示癌症基因组中的新端粒。
02 长读长测序发现 端粒重复序列跨越染色体臂的融合
在探索具有异位端粒重复序列位点的过程中,作者还发现这些重复序列以相对于断点的反向方向存在。长读长测序的数据显示,这些位点很大程度上代表了染色体臂融合事件。通过 PacBio HiFi 和 Nanopore 长读长测序分析候选位点区域,观察到断点后有约 650bp 的反向(CCCTAA)重复序列,以及 chr22q 亚端粒上的 5-8 kb 的序列。覆盖整个事件的单个长读表明,反向(CCCTAA)n重复序列是通过chr22q臂与其短端粒与染色体内位点融合而形成的(Fig 2D)。
Fig.2 通过长读基因组测序揭示癌症基因组中的染色体臂融合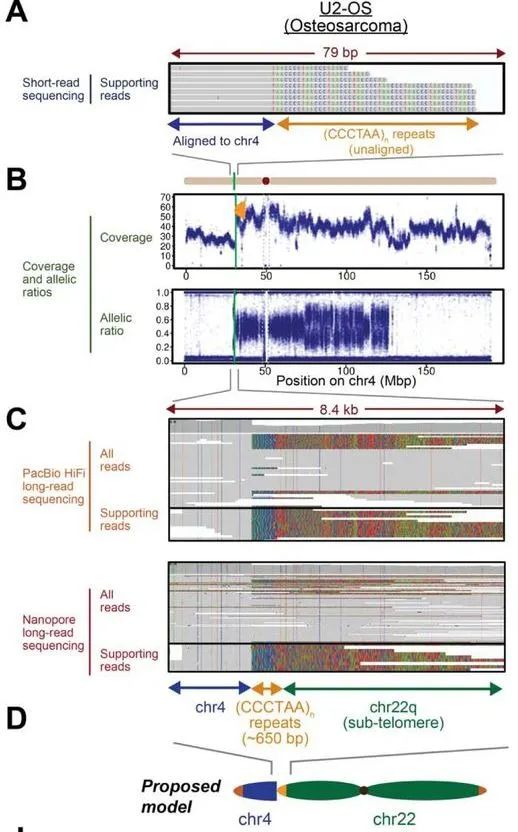
03 新端粒和染色体臂融合事件 可以破坏蛋白质编码基因
端粒序列的插入有可能破坏基因,包括肿瘤抑制因子,从而导致相关的功能影响。例如,作者通过研究发现,一种与免疫治疗反应相关的基因PTPN2,通过长读长测序,在其第一个内含子中发现了新端粒(16 kb),导致第一个外显子和启动子区域出现异常。另外,研究还发现了染色体融合事件破坏基因的现象,包括导致KLF15和FOXN3基因的 5' 区域一半以上丢失的事件。 Fig.3 新端粒和染色体臂融合事件对蛋白质编码基因具有破坏性,并且这一现象在癌细胞系中非常普遍 作者还发现了一些更为复杂的融合事件出现在数个癌症相关的基因中,进而可以确定新端粒和染色体臂融合事件对蛋白质编码基因具有破坏性,并且这一现象在癌细胞系中非常普遍。 这些基因“在癌症中具有重要作用,这也表明新端粒和染色体臂融合对蛋白编码基因的破坏可能会导致肿瘤发生。因此,新端粒和染色体臂融合可能代表了一种重要但尚未得到充分认识的基因破坏机制。” 04 不同长读长测序技术 准确度差异的影响
Fig.4 Nanopore 与 PacBio HiFi测序相比,Sequence divergence差异
在本文中,作者同时使用了PacBio HiFi 以及Oxford Nanopore两种长读长测序技术进行测序。在Nanopore 测序数据中,base calling采用的是 Bonito(版本 0.3.5)和默认的 dna_r9.4.1 碱基检出模型。然而,默认的 Nanopore 碱基识别模型导致数据集中端粒重复序列出现频繁的链特异性碱基识别错误,TTAGG 被误读为 TTAAAA,CCCTAA 被误读为 CTTCTT 和 CCCTGG。相对于PacBio HiFi 测序,两者的 Sequence divergence 呈现了极为明显的差异。 结论 这项研究详细揭示了人类癌症中端粒的维持过程。进一步的癌症基因组长读长测序研究有助于阐明人类基因组高度重复区域的体细胞改变在癌症发病机制中的潜在作用。更广泛地说,长读长测序分析还可以让人们深入了解驱动遗传疾病和进化的染色体重排。
参考文献 Tan, Kar-Tong, et al. "Neotelomeres and Telomere-Spanning Chromosomal Arm Fusions in Cancer Genomes Revealed by Long-Read Sequencing." bioRxiv (2023): 2023-11. 基因有限公司作为PacBio公司中国区合作伙伴,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。 关于基因 基因有限公司成立于1992年,是一家提供生命科学科研仪器、试剂耗材和技术服务的综合服务商。基于“Gene Brightens Every Life • BioTech Connects the World”——“基因燃亮生命 • 生物技术连接世界”的愿景,专注于生命科学领域前沿技术的引进和推广,致力于推动该领域国内科研机构硬件水平及实验方案的革新与升级。同时,公司也一直致力于自主品牌的科研设备的研发与生产,拥有一系列通用性强、互补性高的自主品牌产品。 基因的服务网络遍及全国各地十多个大中城市,拥有包括仪器销售,试剂销售,市场与技术支持,维修,客服,物流等多个部门组成的完整服务体系。 我们希望通过不懈努力,为您的成功铺路搭桥,也为中国的生命科学事业赶超世界先进水平尽一己之力。欲了解更多信息,请访问www.genecompany.cn。
