info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
PacBio HiFi测序助力全基因组变异检测

长读长测序(Long-Read Sequencing,LRS)技术的快速发展,在过去五年内以两种方式根本性地改变了检测基因组变异的格局。首先,NGS测序通常无法发现重复序列区域的结构变异(SV),而长读长测序可以通过直接比对重复序列区域,将SV的检出率提高了3倍以上,其次,LRS使基因组组装变成常规且简单的操作,并且最新的测序技术和组装算法,可以生成完全捕获两种单倍型的分型基因组组装结果。长读长测序的进展大大的提高了我们对突变过程、复发性突变以及与疾病和适应相关的新变异的理解。


现在启动的一些大规模的测序项目,包括 All of Us 和 GREGoR 等,通过LRS揭示了大量和疾病相关的变异。对于这些大规模的测序项目来说,一个关键的问题是如何在最经济有效的LRS数据量(基因组覆盖深度)情况下得到最佳的变异检测结果,并且两个最主流的LRS测序平台,Oxford Nanopore Technologies(ONT)和PacBio HiFi sequencing,在同等的数据覆盖深度下检测变异的情况如何,也是大家都非常关心的问题,今年五月份,美国华盛顿大学的Evan E.Eichler教授团队发表了预印版文章:Whole-genome long-read sequencing downsampling and its effect on variant calling precision and recall,详细研究和评估了ONT和PacBio HiFi平台在不同覆盖度下的遗传变异的检测效果,并得出以下结论:
对于reads-based的方式,ONT和PacBio HiFi在12x覆盖度时,均可以得到良好和稳定的变异检测结果(F1 score大于0.5); Assembly-based(基于基因组组装)可以提升HiFi数据集中SVs和indels检测的准确性和召回率,并且HiFi在基于组装的变异检测上要显著优于ONT(HiFi F1 score更高);
1 基于Reads-based的变异检测
作者使用了DeepVariant和Clair3对单核苷酸变异(SNV)进行分析,在15倍以下的覆盖深度下,PacBio HiFi数据对SNV的召回率(Recall)始终优于ONT,平均优于ONT 0.03。在约10倍的覆盖下,HiFi数据的准确度和召回率达到一个良好的平衡点,准确度达到0.96,召回率达到0.90。在5倍覆盖下,DeepVariant和Clair3在HiFi数据中的表现也均要比ONT数据更优秀(F1值高0.05)。而对于这两个分析软件在准确度和召回率上的表现,DeepVariant在准确度方面表现更好,而Clair3则在召回率方面表现更好。ONT和PacBio HiFi在12x覆盖度时,均能得到良好和稳定的变异检测结果。 图1:不同LRS覆盖深度下SNV,InDel和SV检测的准确度和召回率情况
对于InDels(小于50bp的插入或缺失变异)的检测也呈现相同的趋势,在12x数据覆盖度可以得到一个良好的F1值,但HiFi数据和ONT数据在召回率上差异较大。在所有的测序深度下,虽然ONT数据使用DeepVariant和Clair3的准确度值都较高(平均为0.82),但和PacBio HiFi数据相比,ONT的召回率都明显偏低,在测序层数低于或等于12x时,ONT的召回率相比较HiFi平均低了0.39,而在测序层数高于12x时,ONT的召回率相比较HiFi平均低了约0.31。 而对于结构变异(SVs),两个测序平台对SVs的检测差异最小,HiFi和ONT测序平台之间的F1标准偏差为0.01,可能由于随机抽样偏差,各个软件的召回率均较低,但是PBSV和Delly检测的精度最高,并且这两个软件在低覆盖深度下始终具有高精度(平均为0.89),并随深度的增加保持一致,但在低覆盖深度(低于12x)下的召回率方面,Sniffles表现最佳,平均准确度/召回率/F1值为0.63/0.84/0.71,其次是cuteSV(0.57/0.84/0.67)。 图2:30x和12x情况下,HiFi和ONT平台使用Sniffles对SV检测韦恩图比较。 2 基于组装的变异检测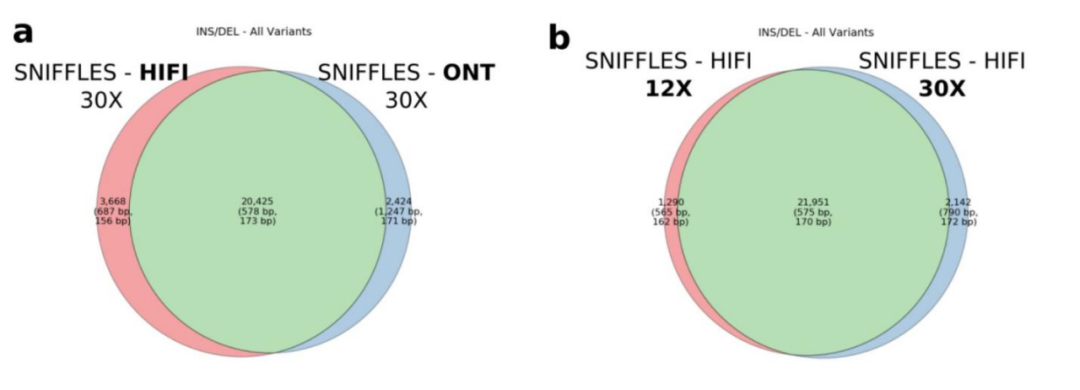
基于组装的方法可以从大的连续单倍型模块中进行变异检测,可以检测更大和更复杂的遗传变异,并为所有形式的遗传变异提供分型。作者使用了三种算法进行了组装:hifiasm、PGAS和Flye。其中hifiasm和PGAS用于组装HiFi数据,而ONT数据使用Flye进行组装,作者提到HiFi数据可以直接组装单倍型分型的基因组,而ONT数据更高的序列错误率并且缺少组装和分型结合的算法,目前基于ONT数据很难获得连续的单倍型分型的组装结果。 对于SNV的检测,基于组装的方法,ONT和ONT超长数据(ultra-long ONT,UL-ONT)均比reads-based的方法显示了较低的精度(平均降低了0.33),在数据量小于12x覆盖深度的时候尤为明显,ONT和UL-ONT的组装结果均显示了大量的假阳性变异,当覆盖深度达到12x时,基于组装的方法ONT和HiFi均显示了优秀的召回率(平均0.96),于reads-based的方法得到的结果类似。作者不推荐用低深度的覆盖率(小于12x)通过组装的方法来检测SNV,不过覆盖深度达到12x或更多时,基于组装方法的SNV检测在准确度和召回率上跟reads-based方法基本类似。 通过组装的方法检测InDels非常具有挑战,PacBio HiFi在高深度(30x)的时候显著降低了检测的假阳性结果,而ONT数据即使在高深度(30x)的时候仍然存在问题,总的来说InDels检测算法的开发和正交验证时LRS技术接下来发展的一个重要领域。 而对于SVs的检测,在高于8x的覆盖深度上,通过组装的方法检测的SVs在召回率上都优于reads-based的方法,至少提高了0.08。 图3: 不同覆盖深度下基于组装方法对变异检测的精度和召回率情况
3 不同数据集和方法比较 作者通过将GRCh38分成1Mbp窗口,与HG00733的HGSVC的SV真实数据集交叉,评估各方法和算法变异检测结果的比较。在低覆盖度(5x)下,reads-based胜过每个组装软件组装的检测。在这些低覆盖深度下,使用HiFi reads的Sniffles表现最佳,在10x及以上的深度下,这种趋势发生了转变,HiFi组装的方法优于所有基于reads-based的算法,最明显的差异发生在15x深度,此时组装后方法比reads-based多检测额外的500 Mbp和383 Mbp的基因组(分别对于插入和缺失)。 作者进一步通过HG002样本比较了这些之前已经发现的有临床相关性的SVs,这些SVs包括273个目标基因或区域,这些基因或区域映射到重复和结构复杂多态性区域。在30x覆盖度下,PBSV能够鉴定97%的这些在临床相关基因中的SVs。在所有技术类型中,在使用PacBio HiFi reads时,仅以8x测序覆盖率报告SV插入和缺失的召回率分别为0.87和0.82。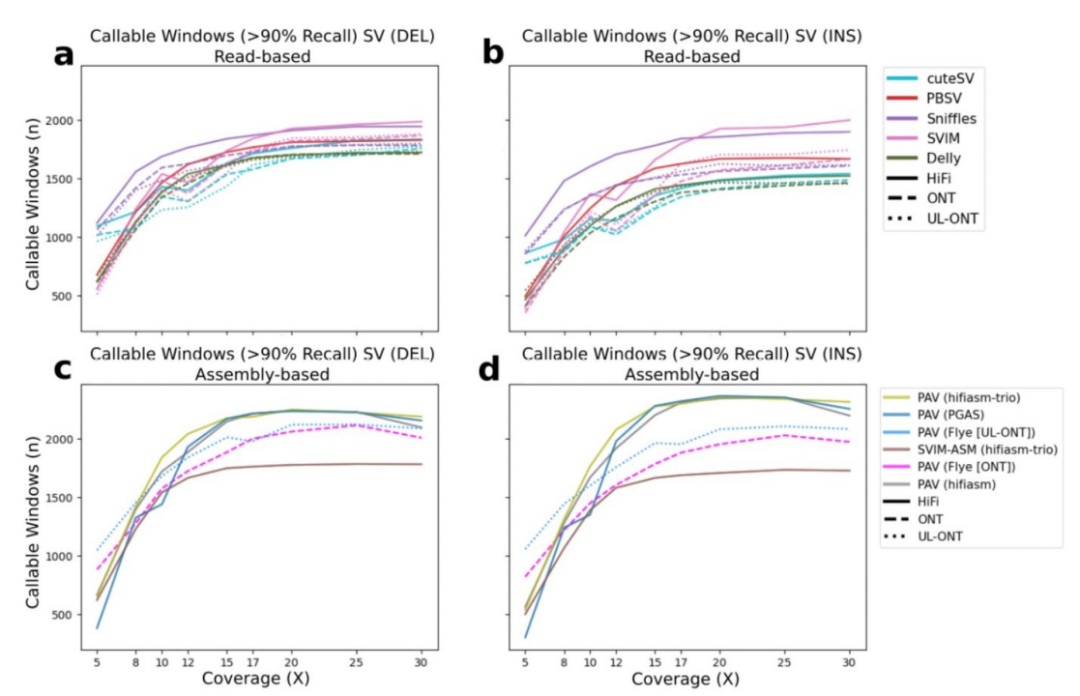
图4: 不同的覆盖深度下SV检测的方法和算法评估
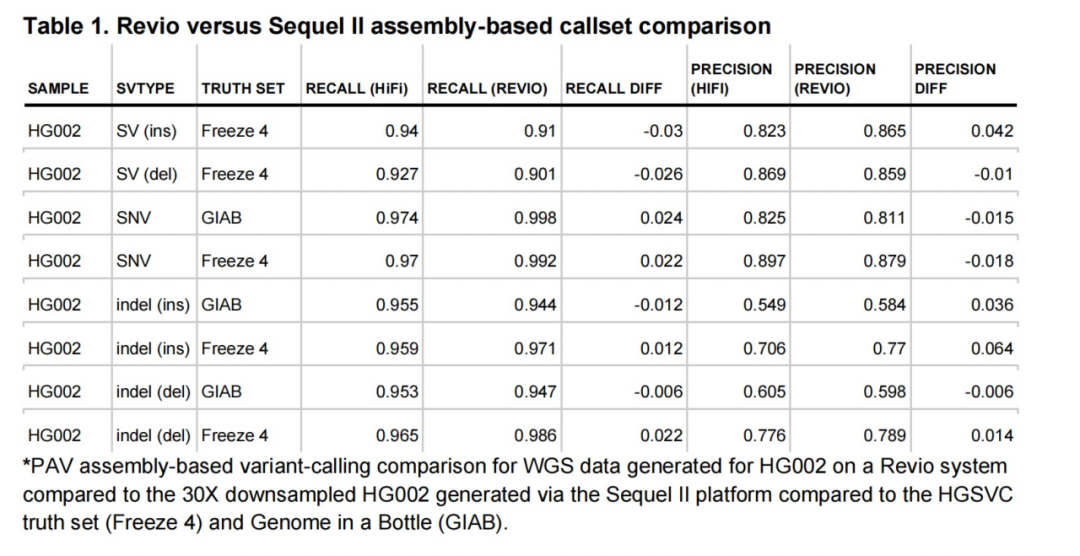
另外作者还评估了一些特殊的复杂区域,如串联重复序列区域,相同多聚序列区域和大结构变异(10kb以上)等,作者发现ONT在相同多聚序列和非相同多聚序列的检测上存在明显差异,即使在高覆盖深度下,相同多聚序列中插入的召回率甚至比整个变异集合低0.10,即使在更高深度下,这些序列类型仍然存在精度较低的情况,即使是30x的reads-based的检测,这些区域的精度也显示了0.06的下降。而对于大的结构变异检测,基于HiFi数据的组装得到了最佳的检测结果。 最后,文章作者使用PacBio最新的Revio测序系统,通过一张Revio的芯片生成了HG002全基因组30x覆盖度的数据量,作者使用hifiasm对Revio产生的HiFi数据进行了组装,得到的组装结果与Sequel II HiFi数据组装的结果进行了比较。连续性(contig N50 = 44 Mbp [Revio] vs. 45 Mbp [Sequel II])几乎一致,而准确性Revio甚至还略高(QV = 57 [Revio] vs. 55 [Sequel II])。基于组装结果的变异检测在召回率(Pearson R = 0.984)和精度(Pearson R = 0.997)方面基本一致,并且在SNV召回率(+0.02 vs. both true stes)和插入片段检测精度(+0.06 vs. HGSVC Freeze 4)方面Revio略有改善。
参考文献 William T Harvey, et al. "Whole-genome long-read sequencing downsampling and its effect on variant calling precision and recall" BioRxiv. 2023.05.04.539448
