info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
美食家眼中的鳝鱼,看PacBio HiFi技术如何来“烹制”

PacBio单分子实时测序(SMRT)技术,以其长读长(长度可达25 kb)、高准确率(≥Q30,即超过99.9%的测序精度)、单分子分辨率、高灵敏度、无GC偏好性等优势,在医学、动植物基因组以及微生物研究中都表现出极大的应用优势。今天,就让我们继续分享PacBio HiFi测序在动物研究领域中的应用。 + + + + + + + + + + + 研究背景 黄鳝,俗称亚洲沼泽鳗鱼或稻田鳗鱼(Monopterus albus),是用于研究性别变化和染色体进化的优秀模式物种。这个物种的低繁殖力(每条鱼大约300个卵)极大地阻碍了其生产和繁殖计划。但是有趣的是,大约3-5%的鳝鱼可以作为雌性存活3年,每条鳝鱼产下超过3000个卵,这被称为非性别逆转(NSR)雌性。 在这里,来自华中农业大学的研究人员提出了一个使用HiFi和Hi-C对NSR雌性基因组进行染色体水平组装的新组合技术。新组装(Mal.V2 _ NSR)数据为838.39 Mb,其contig N50为49.8 Mb ,与之前使用连续长读长测序技术(Mal.V1 _ CLR)获得的组装数据相比,其组装连续性增加了20倍。这一结果不仅提供了性别相关基因和MHC基因的基因组完整版图,还为今后的鳝鱼育种工作提供了很大的帮助,为鳝鱼的遗传和基因组研究提供了有价值的新参考。 + + + + + 
✦+
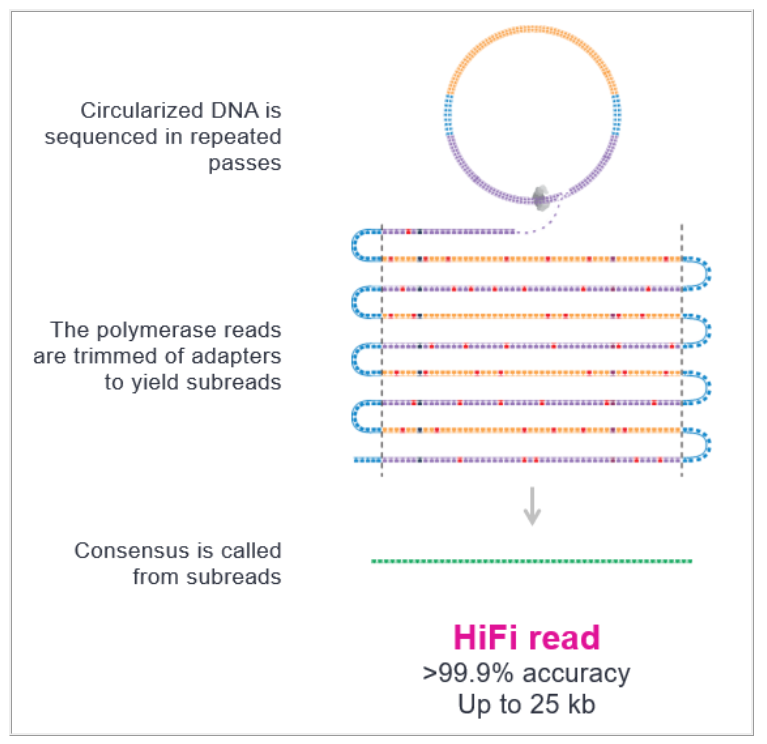
+ 1、基因组组装及评估 在PacBio Sequel II平台上共获得总碱基数为29,369,894,529 bp的1,764,134条HiFi reads,平均reads长度16,648.34 bp, contig N50长度为16,790 bp。使用Hifiasm软件对高准确度的35.4 x 的HiFi reads进行组装,共获得843 Mb的组装数据,包括384个contigs,contig N50为45.38 Mb。 然后,使用从Hi-C文库获得的总共116.59 Gb的clean数据用来搭建contigs,以实现染色体规模的组装。最终,384个contigs被成功锚定到12条染色体上(图1B),并且超过98.27%的contigs在100 kb以上,显示出优异的染色体组装锚定率。最终组装数据为838.39 Mb (图1C) ,contig N50为49.8 Mb,scaffold N50为68.68 Mb,其中包含了38.66 Mb的附加序列(表1)。这个新组装被命名为Mal.V2 _ NSR。 Mal.V2 _ NSR组装结果与Mal.V1 _ CLR高度一致,并且比Mal.V1 _ CLR连续性更好(图1D) ,contig N50值增加了20倍(表1)。同时,借助于Hifiasm组装软件,获得了两套单倍型 (称为 Hap1和 Hap2),Hap1和 Hap2的contig N50分别为14.53 Mb和12.13 Mb (表1)。 图1、(A)鳝鱼图片 (B) Hi-C 组装热图,红块表示序列之间相互作用的强度。(C)基因组特征(Mal.V2 _ NSR)。a,GC 含量; b,基因密度; c,重复; d,LTR; e,LINE 和 f,DNA 转座元件(TE)和RNA转座元件(RNA-TE)。(D)装配连续性改善的可视化。Mal.V1 _ CLR (左)和 Mal.V2 _ NSR (右) 表1. Mal.V2 _ NSR,Hap1,Hap2和 Mal.V1 _ CLR 组装基因组特征的比较 + 2、 基因组注释 在 Mal.V2 _ NSR 数据集中,DNA转座子(21.01%) 、长散在重复序列(LINEs,18.07%)和长末端重复序列(LTR,10.32%)是该组装中重复元件的前三类。总共有47.99% 的基因组包含重复序列,这略高于以前发表的 Mal.V1_CLR组装结果。在Mal.V2_NSR组装中注释了273个miRNA,42,869个tRNA,3,933个rRNA和964个snRNA。最后,共鉴定了22,704个蛋白质编码基因 ,其中共有19,516、 21,813、22,066、20,356和15,457个蛋白质编码基因分别在SwissProt、KEGG、TrEMBL、InterPro和GO这几个数据库中被注释过。 + 3、对比基因组分析 使用全基因组比较工具SyRI ,对新组装数据(Mal.V2 _ NSR)与参考Mal.V1 _ CLR 数据进行了比对,在这两个组装数据中都发现了一个703-707 Mb的共线区域(图2A)。同时,对比两个单倍型的组装数据能够深入了解NSR雌性个体内的单倍型多样性,包括 Hap1中的224个倒位(~3.8 Mb) 、276个易位(~1.75 Mb)和25个重复(~0.22 Mb) ,以及 Hap2中的倒位(~3.8 Mb) 、276个易位(~1.75 Mb)和272个重复(~1.74 Mb) ,占每个单倍型组装序列(66.04和69.64 Mb)的8%左右 (图2B)。此外,对Mal.V2_NSR和大刺鳅M.armatus v1.2(GCF _ 900324485.2)的基因组组装数据进行了同线分析(图2C)。结果表明,M. albus和M.armatus之间共鉴定出176个共线性区块和16,553个基因对。 图2. 基因组比对分析。对比Mal.V1 _ CLR (Ref)组装结果,Mal.V2 _ NSR(Query) 组装结果的同线性和重排位点(SyRI)(图A) 和 Hap1(Query)/Hap2(Ref)的对比图(图B)。(C) Mal.V2 _ NSR 和 M.armatus 之间的染色体保守的同线性(MasArm 1.2)。 + 4、性别相关及主要组织相容性复合 基因的分析 通过使用 Blast 比对nr数据库共发现并证实了59个性别相关基因,并将它们映射到12条染色体上(图3)。23个 MHC 基因被鉴定并分类为 I 类和 II 类 ,并且它们被映射到3,6,9和10号染色体上(图3)。 图3. 性别相关基因和 MHC 基因在新获得组装体(Mal.V2 _ NSR)上的分布。保守的 dmrt1-dmrt3-dmrt2基因簇显示为蓝色,MHC 基因显示为红色,具有两个拷贝的性别基因显示为绿色。 参考文献: Chromosome-Scale, Haplotype-Resolved Genome Assembly of Non-Sex-Reversal Females of Swamp Eel Using High-Fidelity Long Reads and Hi-C Data. doi: 10.3389/fgene.2022.903185 summary 综上所述,作者利用HiFi和Hi-C测序数据进行了染色体尺度的de novo基因组组装并得到了两套单倍型分辨率的组装数据。组装的连续性(contig N50)和基因组质量(碱基准确性和完整性)都高于先前公布的M. albus基因组组装结果,这充分说明了PacBio HiFi reads在de novo基因组组装方面的优势。 HiFi 测序 High Fidelity reads 以HiFi测序为代表的三代测序技术,突破了二代测序GC偏好性及短读长对动植物基因组组装的限制。HiFi reads基于环形一致性测序(Circular Consensus Sequencing,CCS)模式,产生出既有较长的读长(最长可达25kb的长度),又具有很高的序列精度(约99.9%准确率)的测序结果。这一技术对于基因组组装具有重要意义。不仅提高了组装效率、准确率,更重要的是能够实现gap free组装,构建真正意义上的完整基因组图谱。 基因有限公司作为PacBio公司在中国区的代理商,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任,基因有限公司也将一如既往的支持越来越多的PacBio用户。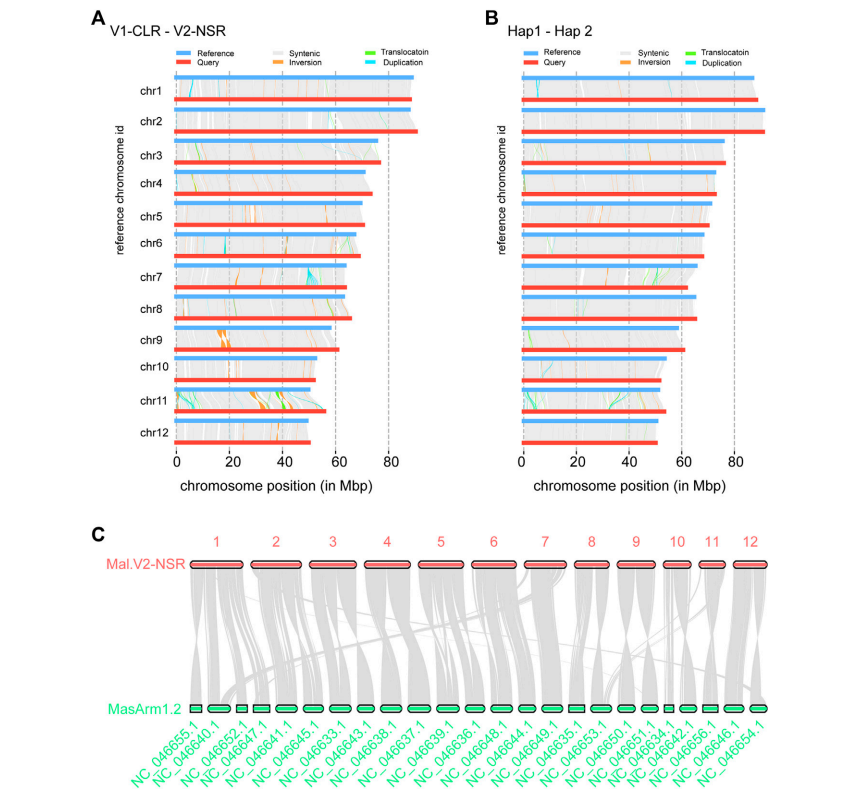

扫描二维码 关注我们 - 获取更多相关信息 -