info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
五大种族SMA患者家系SMN1,SMN2单倍型全面解析

1899年发现第一例脊髓性肌萎缩症(SMA)。它是导致婴儿早期死亡的主要原因,活产婴儿的发病率为1/6,000-10,000,携带者的频率为1/40-80。因其高携带风险以及严重的临床症状,ACMG已建议进行全人群SMA筛查。然而,致病基因SMN1,SMN2所在的基因组区域的复杂问题阻碍了对该疾病的检测。
在《美国人类基因组杂志》(American Journal of Human Genomics)的一篇新论文中,PacBio的科学家与美国基因组医学中心、堪萨斯城儿童慈善会(Children’s Mercy Kansas City)、荷兰人类遗传学系(Department of Human Generics)和Radboud University Medical Center、英国基因组英国有限公司(Genomics-England Ltd.)以及英国基因组研究联合会(Genomic England Research Consortium)合作,重点介绍了Paraphase结合HiFi长读长测序,如何从根本上改变我们研究SMA的方式。

1
复杂的SMN1/SMN2基因区域
在SMA的发展和严重程度中有两个基因发挥重要作用——SMN1和其高度同源的SMN2。这两个基因位于染色体5q13的一段高度复杂的基因组区域,该区域经常发生不等互换和基因转换,导致SMN1和SMN2的拷贝数的变化,SMN1和SMN2的序列几乎完全相同,却有一个功能不同的关键碱基差异(c.840C>T)。在SMN2中,c.840T破坏了剪接增强子,导致7号外显子被跳过,因此,大多数SMN2转录本不稳定且几乎无功能。由于SMN2可以产生少量功能蛋白,因此SMN2的拷贝数是SMA疾病严重程度的调节剂。5q连锁SMA病例中的大多数(〜96%)是由大片段缺失导致的SMN1 c.840C的双等位基因缺失,或者基因转化为c.840T引起的,而较很少部分(〜4%)是由其他导致 SMN1中c.840C缺失的小的致病性变异引起。
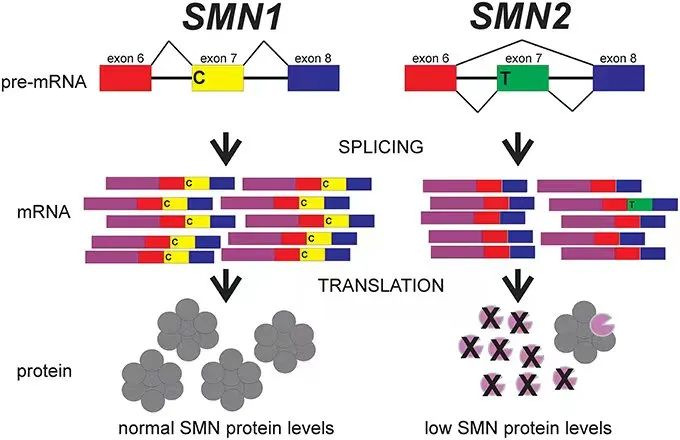
图片来源https://www.frontiersin.org/articles/10.3389/fmolb.2016.00007/full
传统的SMA筛查测试使用基于例如多重连接探针扩增技术(MLPA)和qPCR,确定7号外显子的SMN1拷贝数,以及基于二代测序(NGS)的SMN1检测方法。然而这些方法:
1,不能识别除c.840C>T以外的致病变异
2,传统NGS无法准确分析序列高度相似的基因
3,无法区分等位基因单倍型
区分单倍型对于识别在两个等位基因(1+1)上携带正常SMN1基因的个体与在一条染色体上具有两个SMN1拷贝而在另一条染色体上具有0拷贝的沉默携带者(2+0)之间的差异很重要。沉默携带者约占非洲人口携带者的27%,占其他人口携带者的3%–9%。
“双拷贝SMN1等位基因”意为,具有两个拷贝SMN1的等位基因发生在同一染色体上。先前的研究已经识别到g.32134T>G作为双拷贝SMN1等位基因的标志物,但同样,基于短读长NGS的方法由于序列相似性高而难以区分SMN1和SMN2,也难于识别这些标记。
为了更好地促进SMA筛选,迫切需要一种进行全面的全基因SMN1和SMN2分析的方法。理想情况下,该方法应能够
(1)基于c.840C>T识别完整SMN1和SMN2的拷贝数
(2)识别SMN1中除c.840C>T以外的致病变异
(3)识别沉默型携带者
因而,HiFi长读长测序是解析具有高序列同源性的区域的理想选择,之前在一项针对中国人群的基于扩增子的研究中,已经证明了SMN1中长读长PacBio HiFi测序的实用性。
2
Paraphase
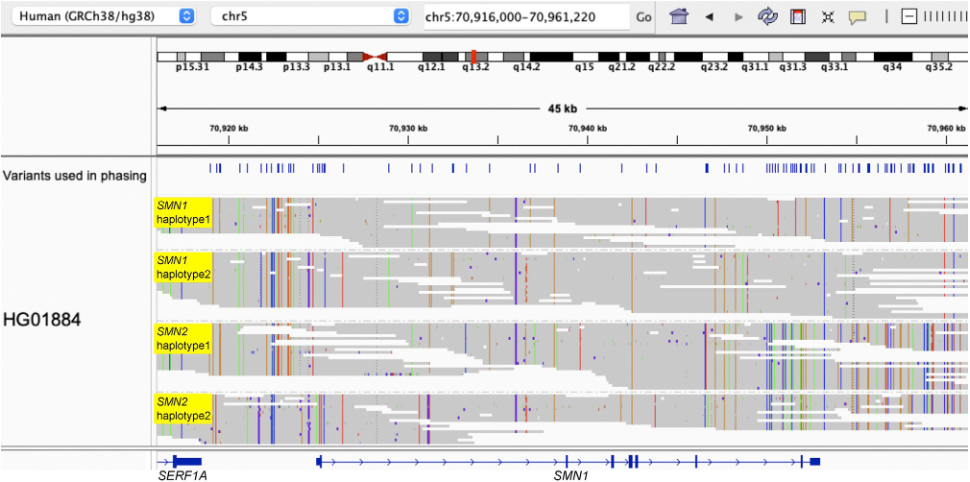
该研究使用最新的Paraphase软件,它基于HiFi数据进行的SMN1和SMN2基因分析。Paraphse提取SMN1或SMN2基因的HiFi 序列,并将其重新比对到SMN1区域。确定了整个44 kb长片段区域(chr5:70917100-70961220,GRCh38)的变异位置,其中包括SMN1基因主体区域及上/下游区域。然后通过连接每个变异位点的区段来组装单倍型(图1)。根据c.840位点的序列将单倍型分配给SMN1或SMN2,即C为SMN1,T为SMN2。此外,Paraphase识别了常见的截短型SMN△7-8,其外显子7-8缺失了6.3 kb。解决了完整的单倍型后,Paraphase通过比对参考基因将变异进行单倍型区分。Paraphase也将单倍型标记为单倍群,以实现进一步的基于单倍型的分析,以鉴定遗传标记。Paraphase可用于全基因组测序(WGS)和基于杂交捕获富集方法的HiFi数据。
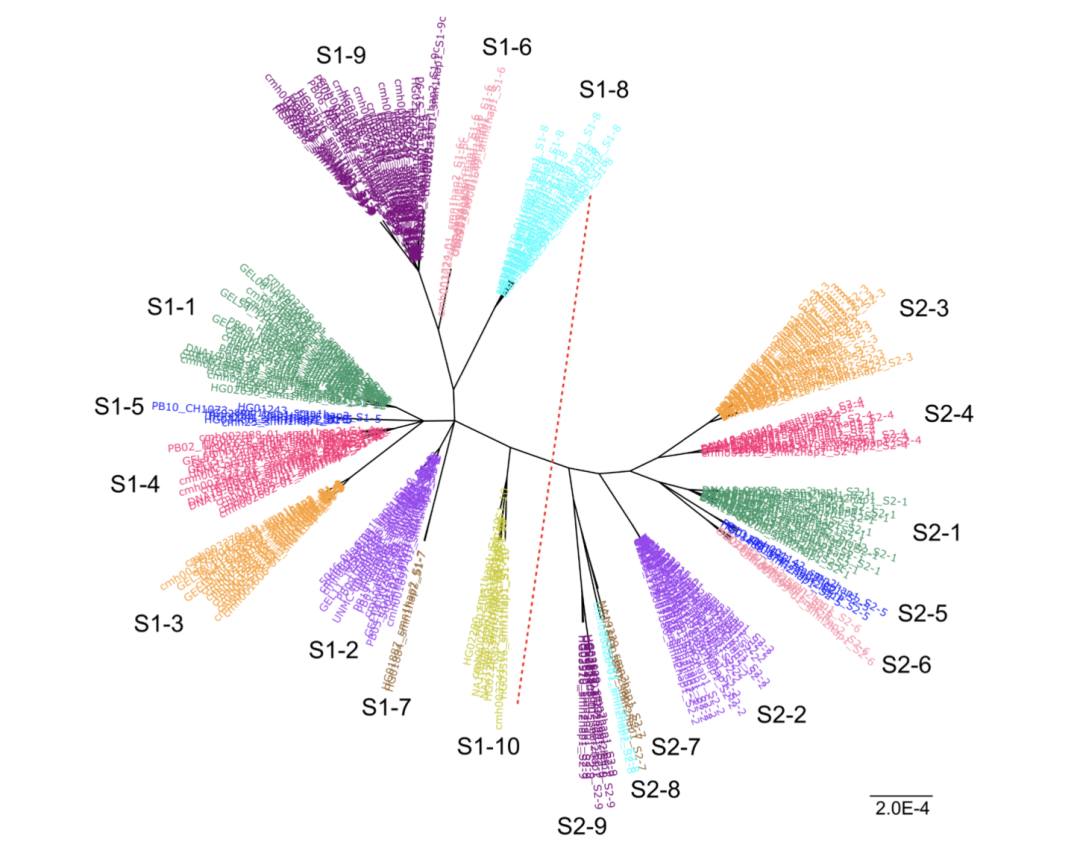
图1.分阶段SMN1和SMN2单倍型 Paraphase产生单倍型标记节段,以便于检查单倍型,并将所有相关读数重新排列到SMN1。 3 来自五个不同种族人群家系测试 在该研究中,纳入了107个Coriell样本,其中7个来自GIAB,100个来自HPRC。还包括了来自Children’s Mercy Kansas City的GA4K的9个携带者(1+0)样本。最后我们纳入了来自 100,000 Genomes Project中的一个SMA 三人家系。有119个具有SMN1 拷贝数信息的样本和116个具有SMN2 拷贝数信息的样本。 人口样本我们包括来自五个种族的341个家系(26个双人家系,308个三人家系和7个四人家系),以研究SMN1和SMN2等位基因的共分离。 将Paraphase得到的SMN1和SMN2 拷贝数结果与,包括基于NGS的WGS、MLPA和SMA Trio推导正交方法进行了比较。SMN1的拷贝数报告一致性为99.2%,SMN2为100%。并识别出了截短型SMN△7-8,其外显子7-8缺失了6.3 kb。 将Paraphase应用于我们收集的人口样本925个SMN1单倍型和645个SMN2单倍型(不包括SMN△7-8)进行了全人群单倍型分析,分别鉴定了10个和9个主要的SMN1和SMN2单倍型。 单拷贝SMN1等位基因 S1-1是所有种族中最常见的单倍型,频率从非洲人的29.9%到东亚人的83.3%不等。S1-2和S1-3在欧洲人,南亚人和混血美国人中也很常见(10%–20%),而在非洲人和东亚人中则不太常见(<3%)。值得注意的是,它不是参考基因组(GRCh38)代表的最常见的单倍型S1-1,而是S1-2。此外,我们观察到了几个非洲特有的单倍群(S1-7,S1-8,S1-9,S1-9d和S1-10)。 图2.全人群单倍型分析确定了主要的SMN1和SMN2单倍型 每个SMN1和SMN2单倍型的基因区域的代表性单倍型序列用于创建无根树。中间的红色虚线分隔SMN1(左)和SMN2(右)。 双拷贝SMN1等位基因 非洲个体约45%–50%的人群携带>2个SMN1拷贝,高于其他种群。双拷贝SMN1等位基因的频率较高,导致2+0沉默携带者的频率较高(估计约占所有携带者的27%),有个体具有两个SMN1拷贝,但都发生在同一染色体上。如果没有谱系信息,使用当前技术不可能直接检测到两个拷贝的SMN1等位基因。 g.27134T>G通常用作双拷贝SMN1等位基因的标记。在我们的数据中,该SNP仅在单倍群S1-8(21.9%),S1-9(100%),S1-9c(100%)和S1-9d(96.3%)中发现。因此它不是双拷贝SMN1等位基因的准确标记。相反,使用HiFi数据,Paraphase可以准确地区分S1-9d或S1-9c与S1-9。此外,能够识别该对的另一个单倍型S1-8,进一步提高了检测双拷贝SMN1等位基因的准确性。 3 序列相似性高的基因拷贝数分析 这一研究对人类基因组中最困难,临床上最重要的区域之一的变异进行了最全面的分析。正如通常所做的那样,除了主要基于c.840C>T的拷贝数测试之外,Paraphase通过对长读长数据的变异位置进行单倍型分析,检测其他致病变异,并能够对沉默携带者进行基于单倍型筛查。可以在捕获或扩增完整的SMN1/SMN2区域时适应于扩增子测序数据。高度准确的HiFi数据可以通过整个基因提供单倍型信息,并且可以轻松地提取大的结构变异,例如SMN△7-8中的6.3 kb缺失和SMN1单倍型S1-9d中的3.6 kb缺失。 在这项研究中,我们对SMN1和SMN2进行了多人群全基因单倍型分析。结合我们的基因水平单倍型和谱系信息,我们确定了形成双拷贝SMN1等位基因的单倍型。最重要的是,确定了一个常见的双拷贝SMN1等位基因,占非洲人双拷贝SMN1等位基因的67.7%。 此项研究仍受限于样本数量,未来使用Paraphse对大量人群的HiFi WGS数据或捕获方法得到的HiFi长读长数据进行分析,将可以更好地表征两个基因的变异,鉴定更多不同的单倍型,分析携带两个或更多SMN1或SMN2拷贝的所有基因,并发现更多用于沉默型携带者检测的遗传标记。 相关文献 Chen X,Harting J,Farrow E,Eberle MA et al.Comprehensive SMN1 and SMN2 profiling for spinal muscular atrophy analysis using long-read PacBio HiFi sequencing. Am J Hum Genet. 2023 Feb 2;110(2):240-250. PacBio 产品仅供研究使用。不用于临床诊断。 基因有限公司作为PacBio公司中国区代理商,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。