info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
NIH百万级人群队列AoU将纳入HiFi技术测定部分数据

1 导读
目前全球有20多个活跃的人类群体基因组计划,这些项目旨在通过对数百万个人类基因组进行测序来建立多样化的健康数据库,以期从精准医学的角度来解析遗传和生活方式对人类健康的影响。准确而全面地识别所有形式的遗传变异及其关联表型特征是了解人类遗传疾病遗传力和起源的关键。遗传力缺失 (遗传力与基因变异关联度低的现象) 被认为与对结构变异 (SV) 检出不足、表型表征不准确,以及关键的罕见变异信息缺失相关。相较于短读长测序,虽然长读长测序在SV和复杂变异检测中表现不俗,但它在大规模人群队列研究中对解析临床相关序列 (例如疾病相关基因) 的贡献未为可知。
来自美国NIH大型基因组项目All of Us (AoU) 的研究团队近日在bioRxiv预印本平台发表了题为“Utility of long-read sequencing for All of Us”的文章,旨在对长读长测序在AoU以及其它大型队列研究项目中的可行性和贡献进行评估。
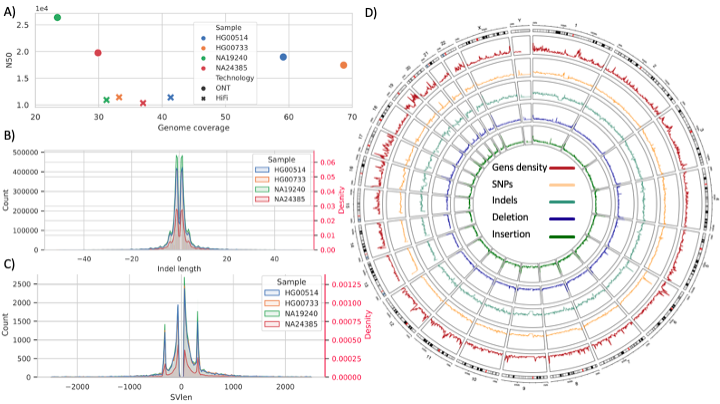
研究者基于来自HapMap和AoU项目的样本比较了短读长测序 (Illumina) 和长读长测序 (Pacific Biosciences HiFi, Oxford Nanopore Technologies) 的关键性能,揭示了不同测序技术在疾病相关基因覆盖度和致病变异检测方面的差异。同时,研究者还阐释了在大型队列研究中使用低覆盖度测序来增加样本数量的优势和挑战。研究结果表明,HiFi数据不论是在小片段变异 (SNV, <50 bp indel) 还是SV检测中均表现最佳。此外,研究者还开发了一个基于云的数据分析流程,该流程优化了长读长数据对SNV,小片段indel和SV的检出性能,为长读长数据在大规模队列研究中的广泛应用奠定了基础。 2 不同测序技术在全基因组中的表现 由于全基因组测序具有随机性,reads的读长以及基因组覆盖度成为了检测遗传变异的决定性因素。基于来自HapMap项目的4个已知遗传变异背景的细胞系 (NA24385, HG00514, HG00733, NA19240),研究者使用不同的变异检测工具 (Longshot, DeepVariant, Clair3, Menta, PbSV, Sniffles) 比较了三种测序数据 (30x Illumina, 35x HiFi, 45x ONT) 对SNV,小片段indel以及SV的检出性能。在该测序深度下,HiFi reads不论是在小片段变异 (SNV, < 50 bp indel) 还是在SV检测中都具有最大F-score值。在SV检测中,HiFi数据的F-score为0.93,ONT为0.91。由于无法识别大的insertion,Illumina的F-score仅为0.45。该结果复现了前人的研究结论,即,长读长测序更适用于SV检测;同时,长读长测序还能实现高准确度的全基因组SNV和小片段indel检出。 图1:A) 长读长数据 (HiFi和ONT) 在HapMap样本 (NA24385, HG00514, HG00733, NA19240) 中的基因组覆盖度 (X轴) 和reads N50 (Y轴) 分布;B) HiFi reads检出的indel长度 (X轴),数量 (Y轴) 和密度 (红色) 分布 (使用DeepVariant + Clair3组合);C) HiFi reads检出的SV长度 (X轴),数量 (Y 轴) 和密度 (红色) 分布 (使用Sniffles + PbSV组合);D) HapMap样本的基因 (棕色),SNV (橙色),indel (浅蓝色),deletion (深蓝色) 和insertion 密度 (绿色)分布。
3 不同测序技术在疾病相关基因集中的表现 一些疾病相关基因具有复杂的重复序列,短读长测序数据对该类基因的变异检测结果往往不甚理想。因此,研究者希望进一步考察长读长测序在疾病相关基因集中的表现。他们使用不同的提取方法分别从T662828295 和T668639440 (来自AoU的两个对照样本) 的白细胞和全血细胞中提取了基因组DNA并进行测序。值得注意的是,该队列的HiFi数据来自HudsonAlpha公布的暨有数据集,其基因组覆盖度仅为6-15x,而Illumina和ONT数据的覆盖度分别为30x和29x。研究者考察了这三种测序技术在以下基因集中的表现:1) ACMG (美国医学遗传学与基因组学学会) 指南列出的73个变异基因 (ACMG 73),包括68个易于测序的基因和5个测序困难的基因;2) Genome in a Bottle (GIAB) 疾病相关基因集 (5,027基因集),包括4,641个易于测序的基因 (4,641基因集) 和386个测序困难的基因 (386基因集)。 三种测序数据在ACMG 73和5,027基因集中都表现良好 (长读长测序略好),但对于386基因集 (测序困难) 的变异检测,长读长测序表现出明显优势。三种测序数据对5,027基因集的变异检出一致性达到 81.33%,该数值在386基因集中下降到60.74%。其中,Illumina reads对386基因集的变异检出性能受到的影响最大,其结果具有高比例的假阳性。 在Illumina reads (30x) 检出的F-score排序前10位的基因中,HiFi reads (8x) 通常会达到同样的或者更高的F-score值,而ONT reads (29x) 由于对3个基因中insertion和SNV的检出失败而表现出较低的召回率。在HiFi reads检出的F-score排序后10位的基因中,有6个基因的HiFi F-score仍然比它们的Illumina F-score高。研究者总结道,利用HiFi reads进行变异检测无论是在精确度还是召回率上均优于其它技术。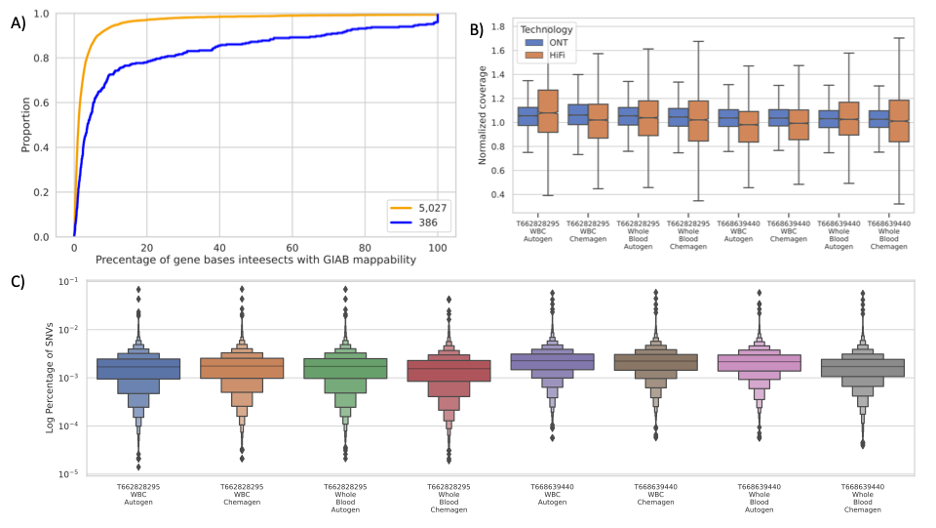
图2:A) 比对困难区域在5,027基因集 (黄色) 和386基因集 (蓝色) 中的碱基百分比;B) HiFi reads (黄色) 和 ONT reads (蓝色) 在AoU样本 (T668639440 和 T662828295) 中的基因覆盖度分布;C) 不同AoU样本中4,641基因集检出的SNV百分比 (取对数)。
4 结论 尽管HiFi reads的覆盖度最低 (6x-8x),但它仍然能够准确检出大多数遗传变异;而Illumina reads虽然具有较高的覆盖度 (30x),但在对SV的检测中表现不尽如意。因此,原始reads的基因组覆盖度或其它简单指标不足以用来评估一项测序技术的实用性。对于长读长测序数据而言,虽然ONT reads具有更大的读长,但具有更高准确度的HiFi reads往往能得到更理想的变异检出结果。因此,更大的读长并不是高灵敏度变异检测的必要前提,reads的高准确度不可或缺。比较三种测序技术的评估结果,研究者认为,长读长测序能够为AoU项目以及其它许多类似规模的队列研究项目建立最完整、最准确的变异数据集。在大规模队列研究中,长读长测序不应该仅仅作为短读长测序的替代品,而应该是研究人员优先选择的方法。 5 展望 长读长测序技术因能“使基因组信息更加完整”而被Nature Methods杂志评为2022年的年度方法。与短读长测序相比,长读长测序能够深入短读长测序难以企及的基因组区域,从而更有效地获得完整的基因组序列,给出更准确的遗传变异检测结果。研究者认为,AoU和其它群体队列项目应该考虑扩大长读长测序的应用规模,同时商榷如何在既定的短读长测序队列中理解和整合由长读长测序鉴定得到的新等位基因的临床相关性。研究者同时写到,“从长远来看,我们可能已经进入到只需要长读长测序的时代,尽管目前还存在测序规模和成本方面的考虑,但不应停下开展更多仅使用长读长测序的队列研究的步伐。” PacBio最新测序平台Revio的上市使HiFi测序在大规模人群队列研究中的广泛应用成为可能。相较于Sequel IIe平台,Revio的通量提升了15倍,一台Revio每年能对1,300个人类全基因组进行测序,能够更有效地帮助研究者获得大规模队列的相关数据并进行解析。同时,得益于Revio平台更低的测序成本,研究者不再需要通过降低测序深度来寻求成本和数据产出的平衡。 6 扩展阅读:All of Us (AoU) 计划 AoU计划 (https://allofus.nih.gov/) 是NIH开展的一项前瞻性队列研究,该计划于2018年5月启动,将囊括100万名参与者的Illumina全基因组测序数据和200万名参与者的阵列数据。AoU的目标是构建多样化的健康信息数据库,该数据库将作为一种战略资源促使基因组测序成为精确诊断和健康风险评估的主要手段,以期更有效地治疗遗传疾病。AoU已经发布了第一批100,000个Illumina全基因组数据集,这些数据集表征了不同族群参与者的基因组变异信息,包括SNV,< 50 bp indel和SV。利用这些数据,AoU旨在通过为不同遗传背景的个人提供预防性和个性化医疗服务从而推动精准医疗的进程。为了获得更高的遗传多样性和更丰富的遗传背景,AoU决定使用HiFi技术对10,000 个人类基因组进行测序,并计划在2023年春季首先发布1,027个HiFi基因组数据集。 相关文献 Mahmoud et al. Utility of long-read sequencing for All of Us. bioRxiv 2023.01.23.525236; doi: https://doi.org/10.1101/2023.01.23.525236