info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
干货:如何利用1000 Genomes数据挑选tagSNP?

一般来说,研究SNP的经典思路/步骤是先找到目的基因的标签SNP(tagSNP),然后挑选功能位点,更后准备样本进行分型验证实验以确定更终有关联意义的位点。
然鹅,自从NCBI宣布龙头数据库Hapmap下线后,如何查询tagSNP呢?
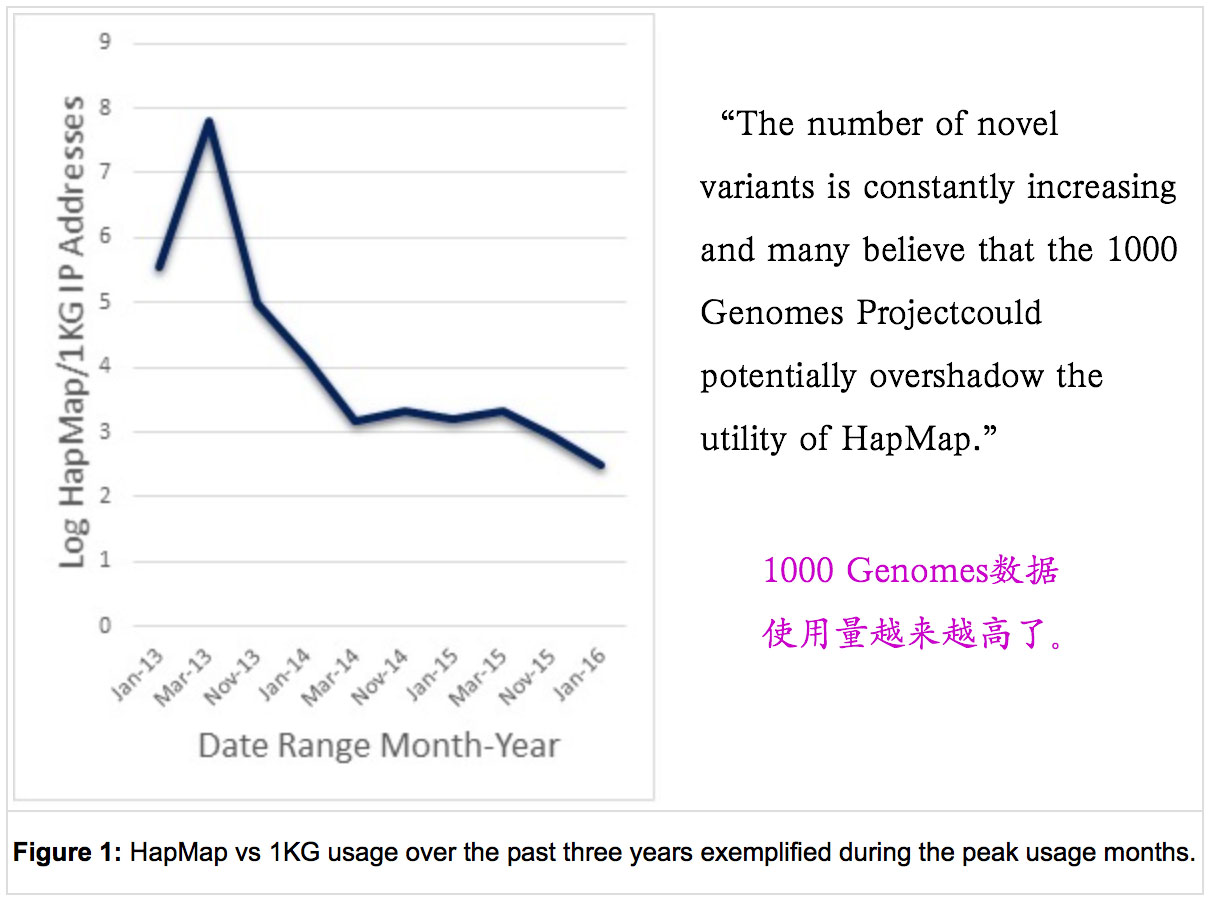
没错,如今得靠1000 Genomes Project数据了,那么下面将介绍如何从一个基因中筛选r2=0.8,MAF>0.05/0.1/0.2的tagSNP位点,以cGAS基因为例:
1. 查询基因的vcf文件
可通过(http://www.internationalgenome.org/about)链接对千人基因组计划进行了解,筛选SNP前需要利用此数据查找基因的Variations,以cGAS基因为例进行介绍:
a. 进入上述千人基因组官网,点击“Browser”按钮,下拉找到“1000 Genomes Project phase 3 browser”,点击进入,即可跳出如下界面:
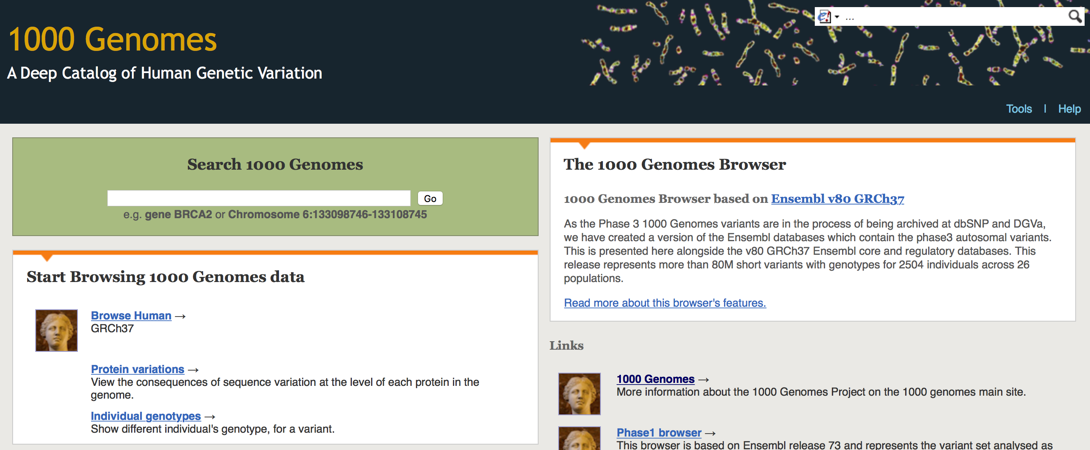
b. 在绿色搜索框区域输入“cGAS”,点击“Go”,跳出如下界面,此时可以看到界面左边的“Get VCF data”功能键为灰色,无法使用
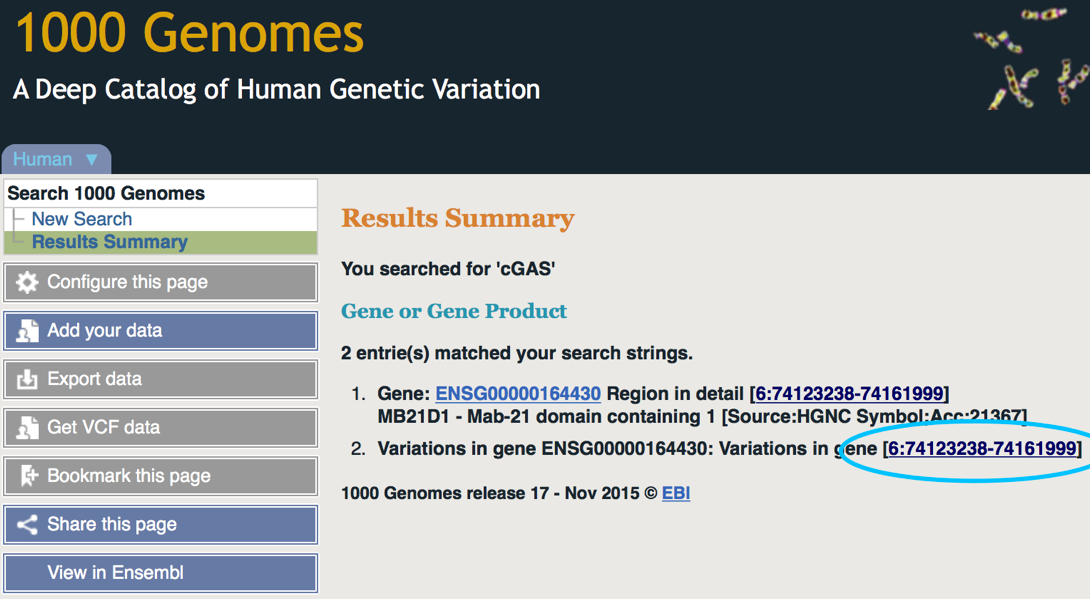
c.点击上图蓝色圈出区域(该区域表示cGAS基因所在染色体位置),跳出界面:
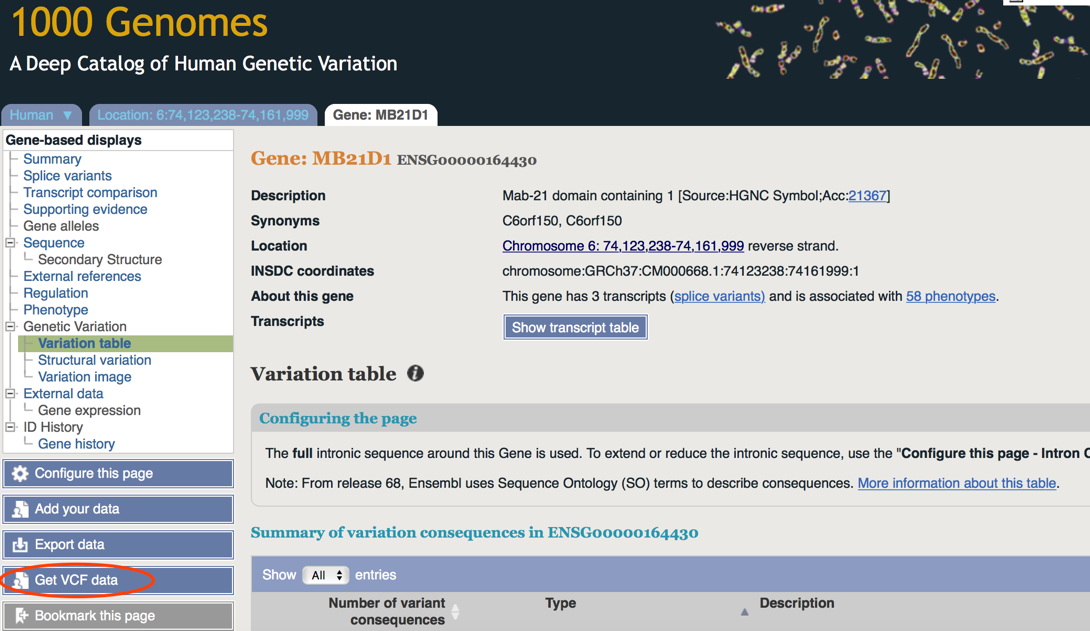
d.弹出如下个界面,下拉点击“Next”,再弹出第二个界面,右键图中链接即可保存该基因的vcf文件,文件名后缀为“vcf.gz”。
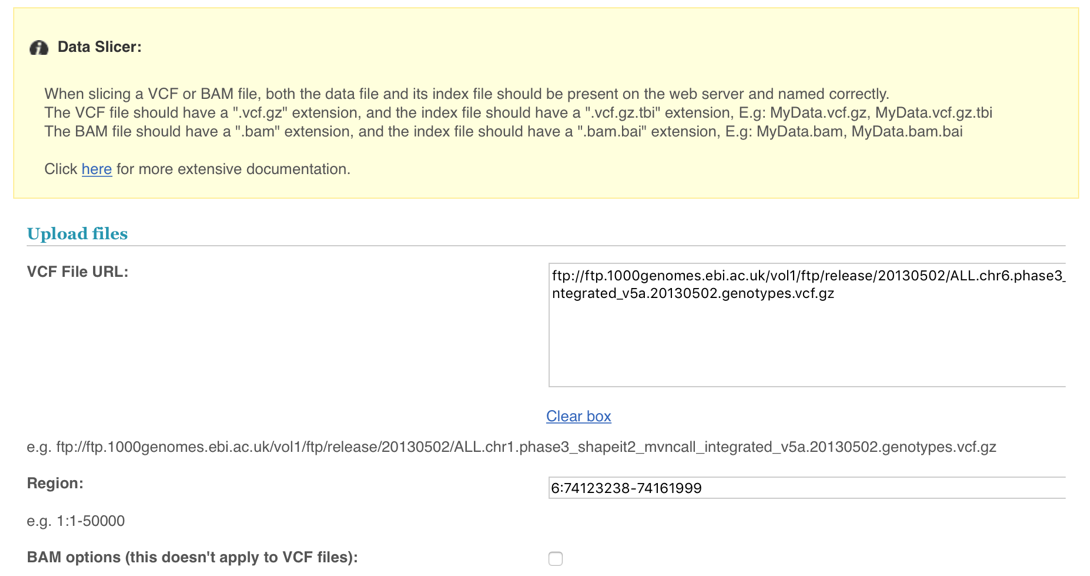
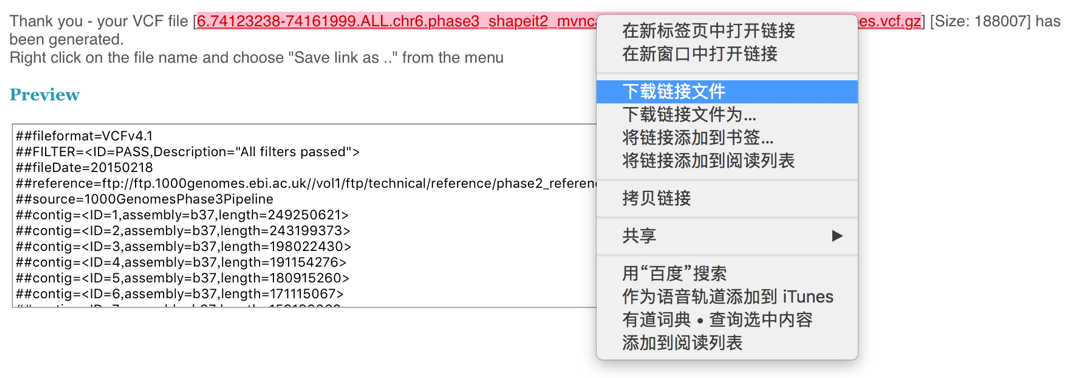
2. 转换文件格式(vcf to ped)
由于筛选tagSNP的软件Haploview只能识别ped格式的文件,故必须将查询到的vcf文件转换成ped格式,可使用在线工具(http://grch37.ensembl.org/Homo_sapiens/Tools/VcftoPed?db=core)
a.进入网站,可以看到如下界面,输入参数,“Name for this Job”可直接输入研究的基因名“cGAS”,“Region Lookup”输入该基因所在染色体区域,选择populations(CHB代表中国北京汉族人群、CHS代表中国南方汉族人)
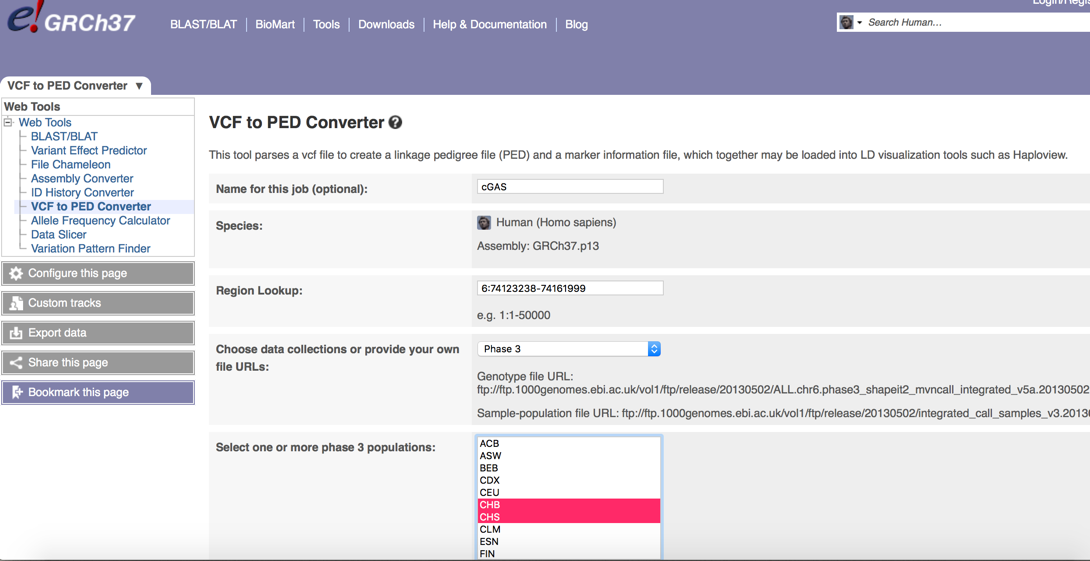
b. 其他参数不动,下拉界面点击“Run”,会出现下图
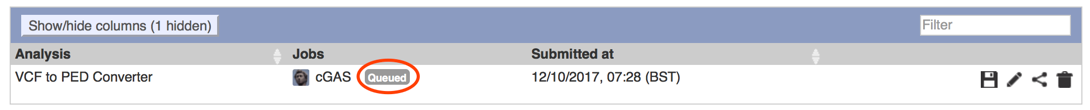
c. 完成后红色圈出部位会由“Queued”变成“Done”

d. 点击旁边的“View results”,跳转下图,下载保存两份文件即可,文件名后缀分别是“.info”和“.ped”
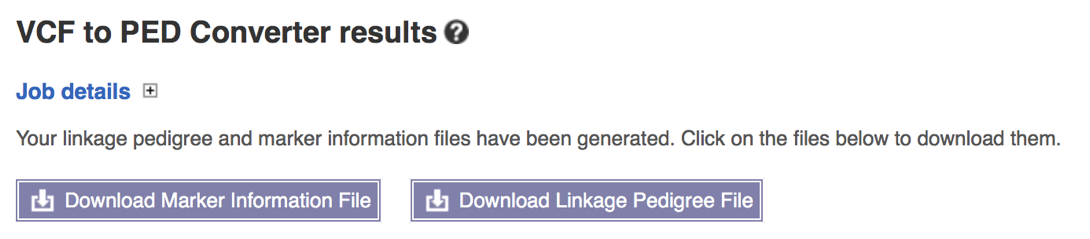
发现没有,其实只要记住基因在染色体上的位置就能通过第2步直接生成ped文件了。当然也可以在NCBI数据库上查找该数据,注意必须选用GRCh37版本的,这样才能与1000 Genomes数据对应。
二、Haploview软件安装
Haploview软件是做SNP研究必备的一个工具,除了下面要介绍的tagSNP选择外,还可做case-control association study and TDT分析、LD block的制作和分析等等。
注:Haploview软件必须在java环境下运行,故必须先安装Java
三、tagSNP筛选
a. 打开Haploview软件,点击“File”-“open new data”,弹出如下界面
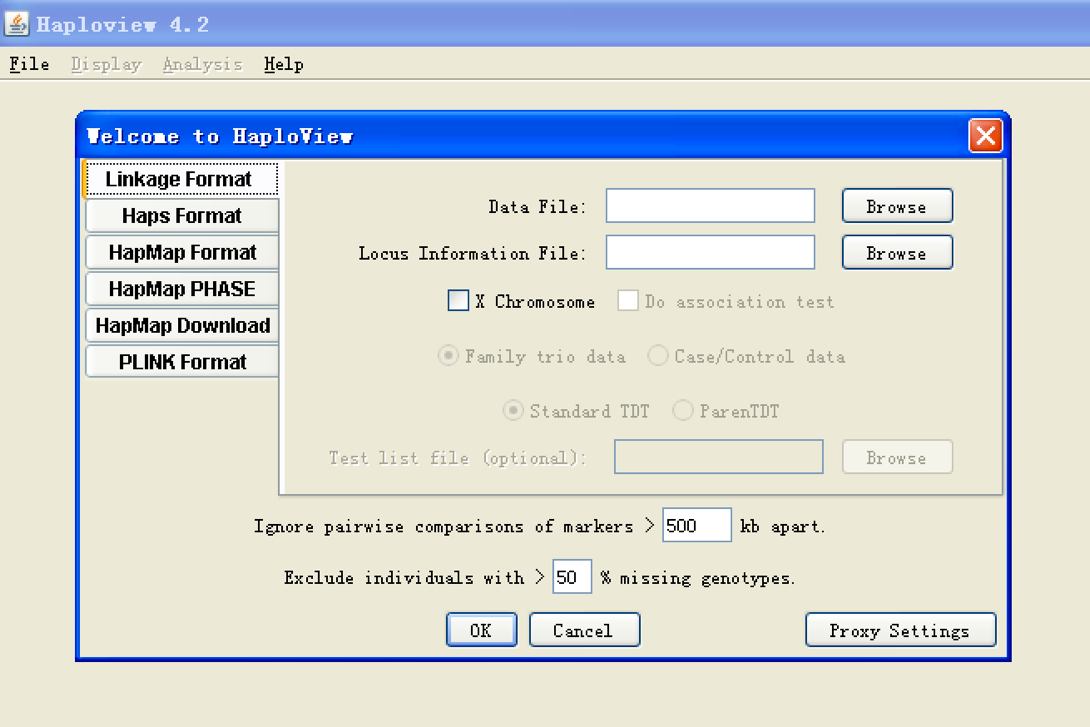
b. 选择“Linkage Format”-“Data File”点击Browser选择之前保存的ped文件,“Locus Information File”点击Browser选择info文件
注:“Ignore pair comparisons of markers”一般填写500kb,即忽略距离大于500kb的两个SNP的haplotype,因为即使算出来在遗传学上也没有很大的意义。
c. 点击“OK”,即可进入下图“Check Markers”选项界面:

d.上述界面中红色圈出区域“HW p-value”为默认值为0.001,“Minimum minor alleles freq.”可设置为0.05/0.1/0.2(根据要求设置),点击“Rescore markers”,然后点击界面上端的“Tagger”按钮,跳转如下界面:
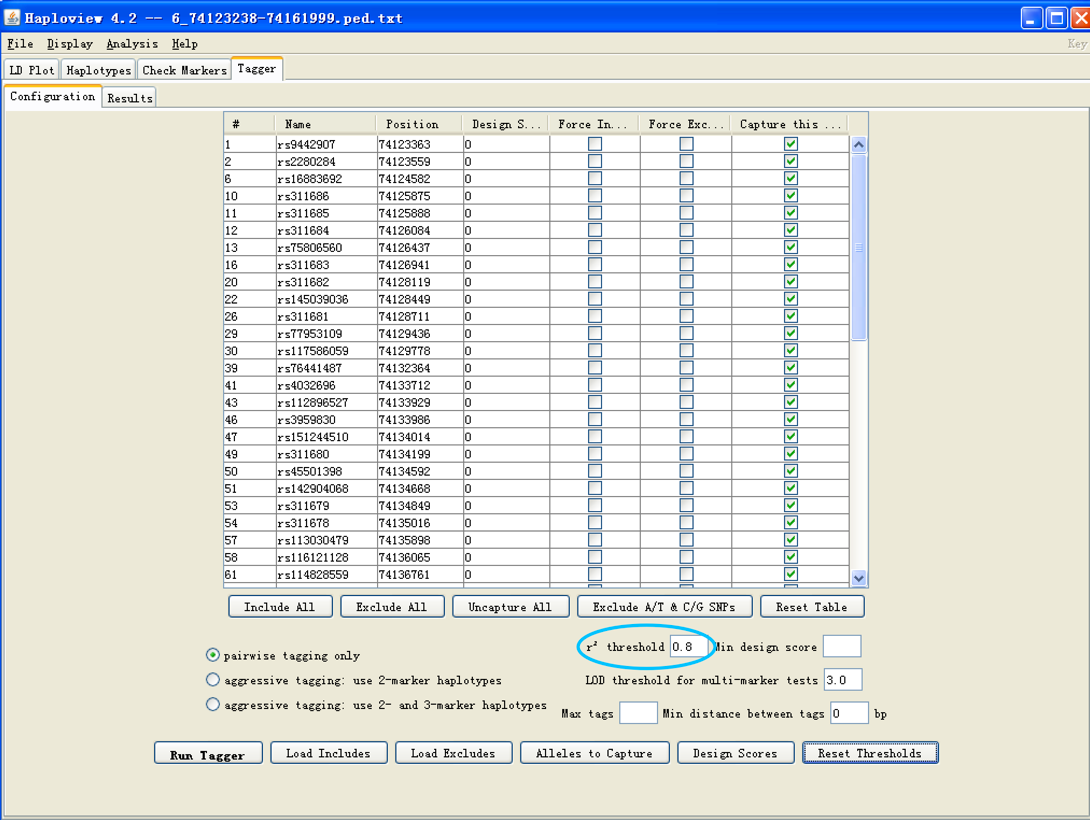
e. 上图蓝色圈出部位“r2 threshold”可设置为0.8(根据需要,亦可设置为1),点击“Run tagger”
,即跳转如下Result界面,表示在r2为0.8,MAF为0.05条件下,cGAS基因的tagSNP有28个,点击“Dump Tags File”即可保存位点rs ID,保存时可在文件名后面加上“.txt”。
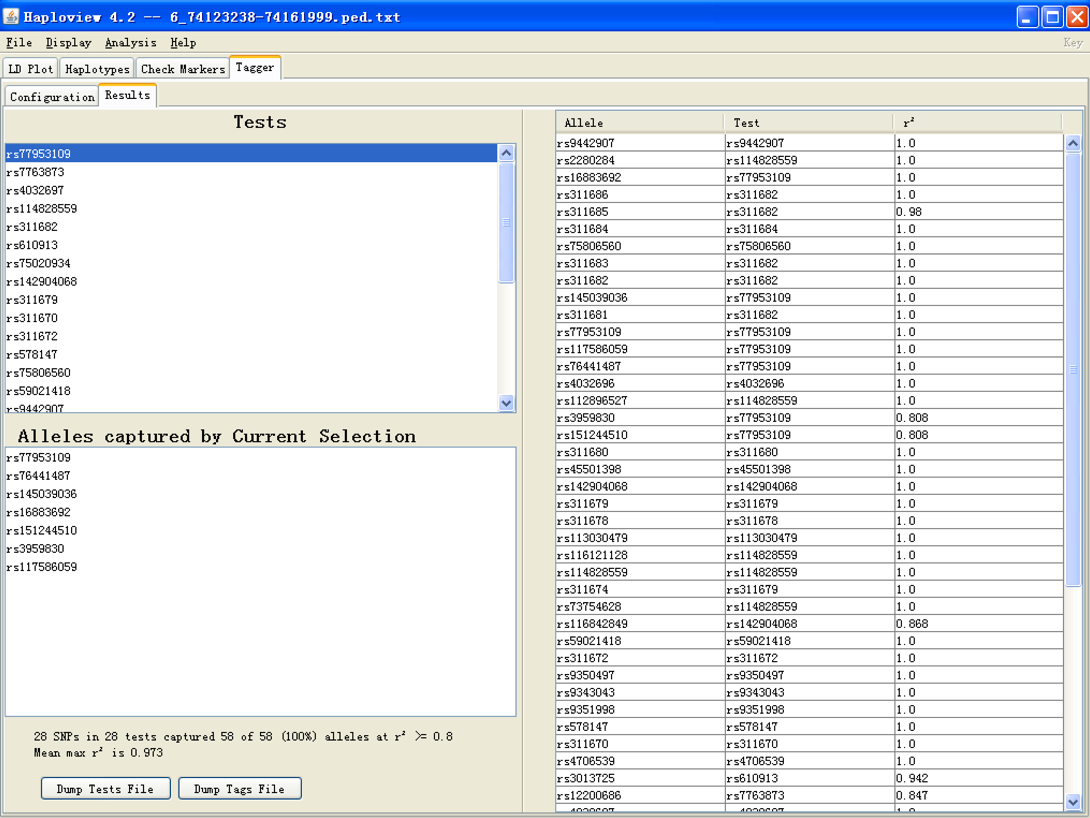
以上操作就能查找出某个基因的tagSNP了~
当然,即便按照上述这么详细的教程来操作,可能还是会有小伙伴们在中途遇到各种errors,小编会在下一期整理一些Trouble Shooting/FAQ供大家学习,记得关注“基因公司”官网哦~
往期链接:
技术负责人:
成小姐:18817595251
Shirley_cl@baygene.com