info@genecompany.com|
基因快讯|


技术交流

扫描二维码
或添加“GeneGroup003”
获取更多更新资讯
商城订购

扫描二维码
或添加“基因商城(GeneMart)”
手机下单,快人一步
售后服务

扫描二维码
或添加“GeneGroup005”
获取更快速售后支持
基于HiFi测序打造首个图形化的绵羊泛基因组图谱

泛基因组(Pan-genome),是指存在于某一物种中的全部基因序列。各种作物,畜牧动物在长期的驯化过程中,不同品系间都形成了各自独特的遗传性状。发现和利用遗传变异是作物和畜牧动物繁育改良的关键。
PacBio HiFi 测序技术的出现,基因组组装不再消耗大量的人力物力,往往在数天之内即可完成高质量的基因组组装。当这一优势应用于泛基因组的研究与应用时,为泛基因组带来了飞速的发展,可谓是泛基因组时代的引擎。
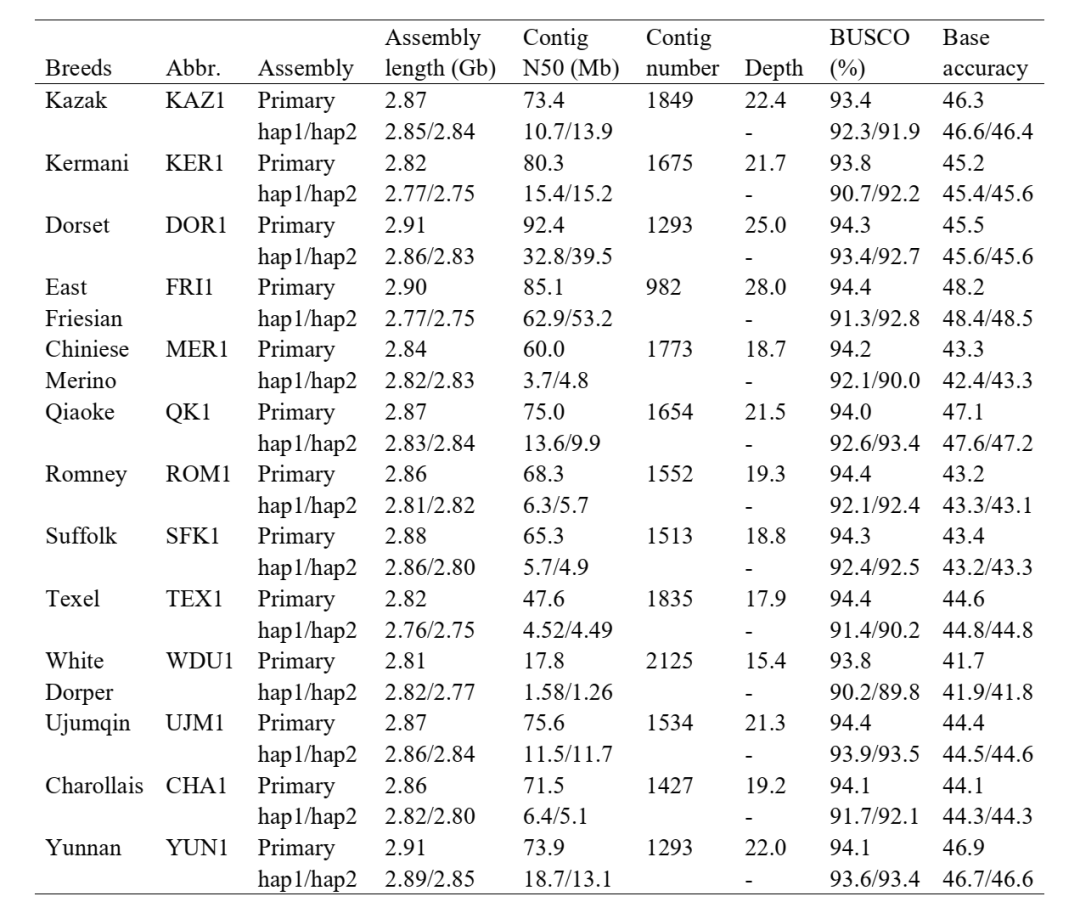
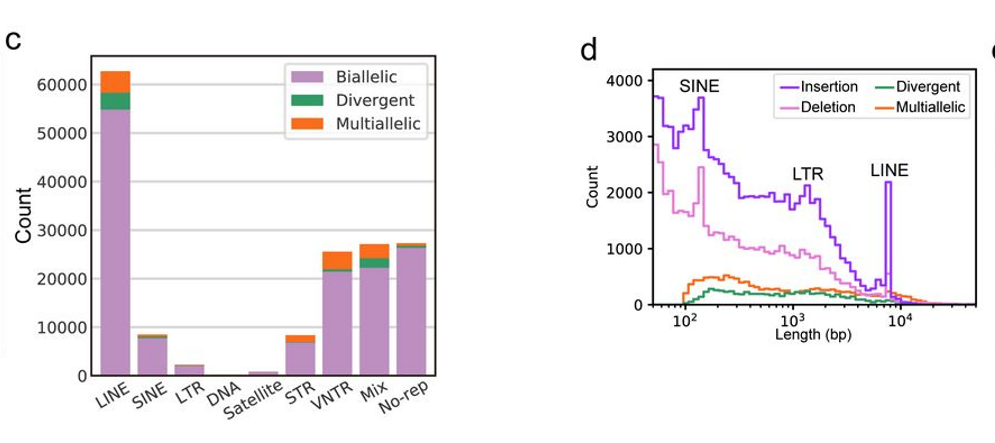
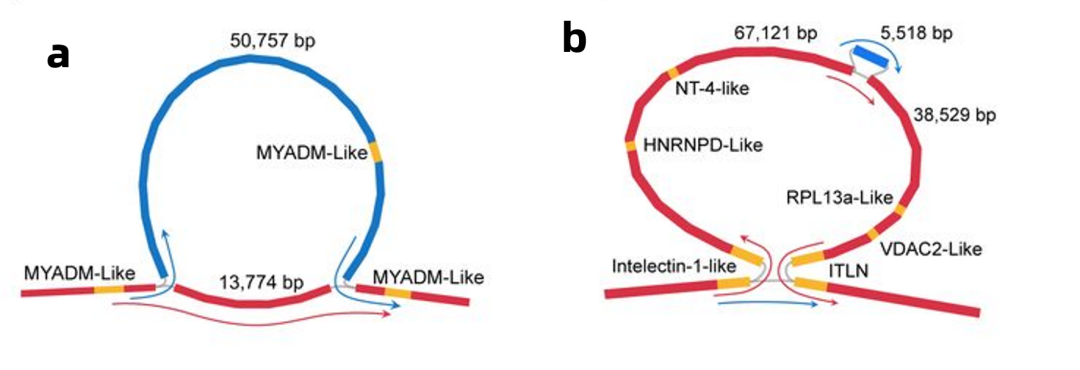
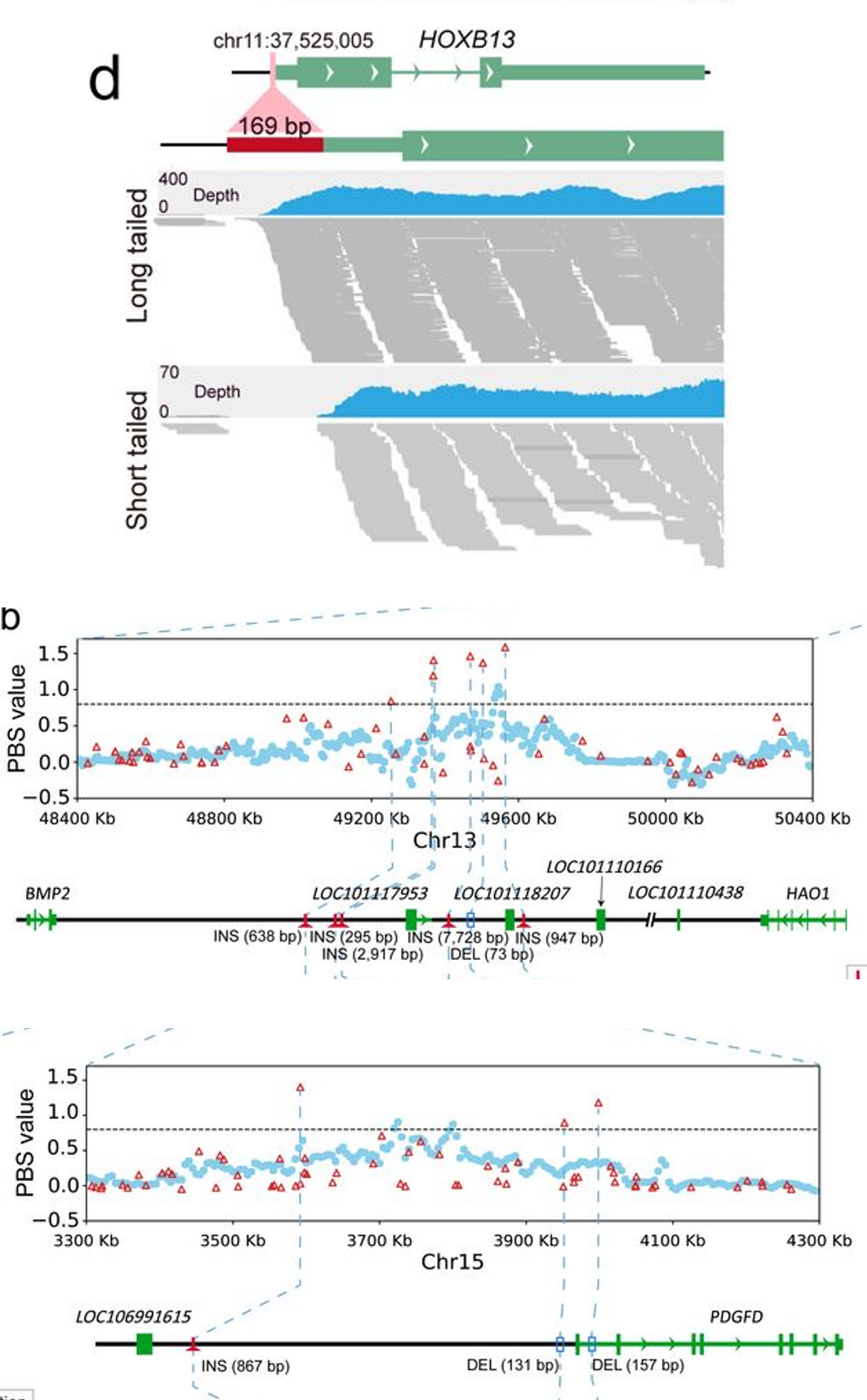
作为国内研究畜牧动物基因组的重要科研团队,西北农林科技大学的姜雨教授团队于2021年在动物遗传育种领域Top期刊Genetics Selection Evolution发表了首个萨能奶山羊的高质量参考基因组Saanen_v1的文章1,该研究利用高深度Pacbio测序和Hi-C辅助组装技术,其Contig N50达到了46Mb。 而近期,姜雨教授团队,以及新疆农垦科学院畜牧兽医研究所甘尚权团队,对绵羊泛基因组进行了研究,构建了首个图形化的绵羊泛基因组图谱,并揭示了相关的结构变异及其与绵羊尾部表型的相关性。该研究的相关文章已在预印本网站bioRxiv上发表2。 13个绵羊品种,高质量的单倍型基因组组装 在这一研究中,作者采用PacBio HiFi测序技术,对来自欧洲,亚洲,中东以及非洲的共13个绵羊品种进行了全基因组测序,每个样本采用2-3张SMRT Cell 8M芯片进行HiFi测序,并采用hifiasm流程进行了单倍型的参考级的基因组组装。每一个品种的单倍型组装结果如下表所示。 表1:13个绵羊品种的基因组单倍型组装汇总 其中,组装最为完整的FRI1基因组,代表组装连续性的Contig N50已高达85.1 Mb,整体平均的Contig N50也达到了68.2 Mb,这一组装的质量已经远高于2021年发表的绵羊基因组参考序列2ARS-UI_Ramb_v2.0。ARS-UI_Ramb_v2.0这一参考基因组正式发布于2021年,虽然相较于2017年美国贝勒医学院所发布的基因组4已经有了很大的提升。但不可否认,当HiFi测序技术推出后,不仅更进一步提升了基因组组装质量,并使得快速高质量的基因组组装得以实现,以至于开启了绵羊泛基因组时代的引擎。 表2:近期发布的绵羊基因组的组装比较2,3(编者整理) 泛基因组构建及变异的分析 随着13个品种,共26套高质量的单倍型基因组的构建,作者进一步分析了结构变异,并基于结构变异,单碱基多态性构建了绵羊基于图形的复杂泛基因组。以已有的绵羊参考基因组ARS-UI_Ramb_v2.0为骨架,将26个单倍型的基因组数据进行回填比对。通过这样所获得的序列变异相较于简单的contig分析来说丰富得多,也更适宜于发现复杂的变异信息。通过对变异进行分类,发现大多数与LINEs相关,其次是VNTR以及与分散的重复相混合的变异。 图1:泛基因组中变异分类以及相关变异的长度 通过分析,分类为 SINES的变异集中在150 bp 大小,LTR相关变异集中在1,300 bp左右,较大的LINE相关变异为 7,750 bp(图 1)。 通过预测,插入变异囊括了 588 个基因,而缺失变异则可能影响了 1920 个基因,包括其外显子区域。非参考序列的不同等位基因序列则包含了 31 个基因。作者揭示了一个包含 MYADM -like的重复序列的发散位点的完整序列,长达50 kb(图 2a),此前有报道称其与平均红细胞血红蛋白浓度性状和断奶羔羊的体重有关,但却未得到过序列信息。最大的多等位基因区域的一个代表性缺失高达 105.6 kb,但也发现其中嵌套了 5.5 kb 的插入序列(图2b)。 图2:图形化展示泛基因组中结构变异的插入与缺失 与绵羊尾部性状相关的基因及其变异 在后续对结构变异的分析中,作者还进一步发现了与绵羊尾部性状高度相关的基因及其变异,包括HOXB13基因中影响其表达的一个169 bp的插入片段,以及BMP2,HAO1,PDGFD中的结构变异和单碱基变异。而PDGFD中,插入变异片段可达867 bp;BMP2,HAO1所在的IBH区域,最大的插入/缺失变异可达7.7 kb。 图3 与绵羊尾部性状相关的基因中存在的结构变异 相较于重测序,图形化泛基因组研究的意义深远 对不同品种的基因组结构变异进行全基因组分析对于了解遗传多样性的全部内容以及查明它们与表型性状的关联至关重要。过去,这一分析往往依赖基于NGS的重测序完成,一般将短读长的NGS数据映射到线性参考基因组中,这导致了无法发现具有明显结构变化的区域。基于图形的泛基因组模型在很大程度上重塑了评估序列多样性整体格局的方法,不再局限于线性的参考基因组信息,采用高质量的组装进行比对,更进一步在单倍型的条件下挖掘结构变异等显著差异的区域。这一方法上的改进可能进一步改变基因组研究领域的现状。 PacBio HiFi 测序能够在短时间内实现13个样本的单倍型组装分析,为高质量的泛基因组构建夯实了基础。也更进一步为功能基因组的分析提供了坚实的数据支撑。 相关文献 Li, Ran, et al. "A near complete genome for goat genetic and genomic research." Genetics Selection Evolution 53.1 (2021): 1-17. Li, Ran, et al. "The first sheep graph pan-genome reveals the spectrum of structural variations and their effects on different tail phenotypes." bioRxiv (2021). Davenport, Kimberly M., et al. "An improved ovine reference genome assembly to facilitate in-depth functional annotation of the sheep genome." GigaScience 11 (2022). Oar_rambouillet_v1.0, https://www.ncbi.nlm.nih.gov/assembly/GCF_002742125.1/ 长按并识别左边二维码,添加企业微信联系人HiFi reads,了解PacBio的最新资讯,还可以与我们取得在线的联系。 基因有限公司作为PacBio公司在中国区的独家代理商,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。